Nature Chemical Biology丨陈飞团队与合作者揭示INTAC复合物通过表观沉默介导BET抑制剂肿瘤敏感性的新机制
染色质调节和转录因子能够通过改变表观遗传修饰与转录活性,调控基因表达,决定细胞命运。溴结构域和额外末端结构域(bromodomain and extraterminal domain,BET)蛋白是调控基因转录的重要蛋白家族,其作用方式是识别并结合乙酰化的赖氨酸位点,招募染色质调节和转录因子,激活基因转录;同时,BET蛋白直接参与RNA聚合酶II(RNA Polymerase II, Pol II)介导的转录进程,促进转录延伸。研究发现,BET蛋白对于调控肿瘤细胞生长和存活具有重要作用,已成为癌症治疗的重要靶点。随着BET抑制剂的开发和应用,这些癌基因驱动的肿瘤治疗有了新的希望。然而,BET抑制剂的疗效通常会受到多种原发或者获得性耐药的限制,严重阻碍其在临床中的应用。因此,探索肿瘤细胞对BET抑制剂耐药的机制对于改善BET抑制剂的疗效,推动其在临床中的应用具有重要意义【1,2】。
在高等生物中,转录机器的激活或抑制受到严格的分子调控。近年来,INTAC复合物被发现是调控转录机器命运的核心因子。该复合物由RNA内切酶复合物Integrator和磷酸酶PP2A的核心酶(PP2A-AC)共同组成,并包含多个结构和功能模块:骨架模块、肩部模块、RNA内切酶模块、磷酸酶模块、SOSS模块以及辅助模块。其中,两个酶活模块分别发挥不同的作用机制:RNA核酸内切酶模块除对snRNA终止起到关键作用外,还负责蛋白编码基因的Pol II启动子近端早期终止;而磷酸酶模块负责阻止Pol II的暂停释放,抑制基因转录。对于辅助模块,尽管一些研究表明其在 INTAC 内切酶介导的 snRNA 3’端的加工过程中起辅助作用,其丧失会导致轻微的 snRNA 终止缺陷,但该模块的功能仍未被完全了解【3-6】。
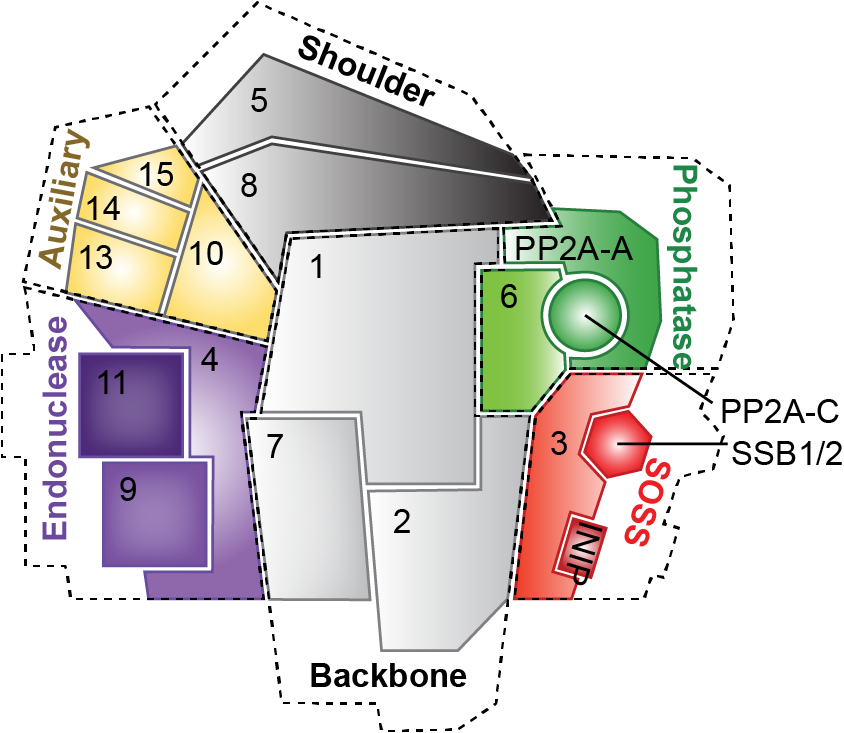
图注:INTAC各模块亚基模式图
2025年1月14日,复旦大学生物医学研究院陈飞课题组、中国科学院分子细胞科学卓越创新中心/杭州高等研究院姜海课题组以及同济大学附属上海市肺科医院张鹏课题组合作,在Nature Chemical Biology期刊在线发表题为Catalytic-independent functions of Integrator-PP2A complex (INTAC) confer sensitivity to BET inhibition的研究论文。该研究首次发现INTAC辅助模块能够以非酶活依赖的方式调控基因转录。辅助模块直接招募RACK7-KDM5C复合物,降低组蛋白H3第四位赖氨酸(H3K4)的甲基化修饰水平,抑制肿瘤细胞生长相关基因的表达,介导肿瘤细胞对BET抑制剂的敏感性。

研究者通过全基因组CRISPR筛选发现,靶向INTAC辅助模块亚基Ints10、Ints13、Ints14和Ints15基因的sgRNA在BET抑制剂JQ1处理的耐药细胞中富集程度最高,而INTAC其他模块亚基却没有明显的富集,提示INTAC辅助模块可能具有相对独立的功能,能够以非酶活依赖的方式介导肿瘤细胞对BET抑制剂的敏感性,随后基因敲除和功能验证的结果也证实了我们这一假设。
进一步的研究发现,INTAC辅助模块能够通过分子间直接接触招募组蛋白去甲基化酶复合物RACK7-KDM5C到染色质的启动子区域,通过降低染色质上组蛋白H3K4甲基化修饰,在整体上影响基因的转录,尤其是对于BET蛋白调控的包括细胞周期、DNA复制等通路在内的相关基因的转录。因此,对于INTAC辅助模块缺失或者低表达的耐药肿瘤细胞,联合应用组蛋白H3K4甲基化转移酶抑制剂MS67(靶向降解COMPASS复合物关键亚基WDR5的小分子药物),可以有效改善肿瘤细胞对BET抑制剂的耐药。
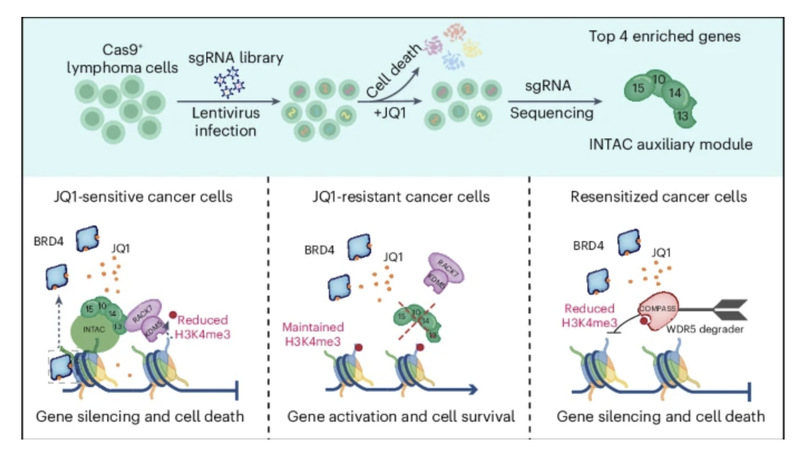
图注:INTAC辅助模块工作模型。通过全基因组CRISPR筛选,发现靶向INTAC辅助亚基的sgRNA是耐药细胞中富集程度最高的。敏感肿瘤细胞中,BET抑制剂作用下,INTAC辅助亚基能够直接招募RACK7-KDM5C复合物,降低组蛋白H3K4me3修饰,抑制基因转录,导致细胞死亡;耐药肿瘤细胞中,INTAC辅助亚基缺失,不能招募RACK7-KDM5C复合物到染色质上,组蛋白H3K4me3修饰维持在正常水平,基因转录激活,细胞存活;耐药肿瘤细胞中联合应用MS67,能降低组蛋白H3K4me3修饰,重新沉默基因转录,细胞死亡。
综上所述,该研究论文首次发现了INTAC辅助模块具有独特的非酶活依赖功能,能够介导肿瘤细胞对BET抑制剂的敏感性,对于改善BET抑制剂在临床应用中的耐药问题提供了新的思路和解决方案。
值得一提的是,自2020年首次发现INTAC复合物以来,陈飞课题组围绕INTAC复合物进行了多方面的研究,首次报道了INTAC通过其磷酸酶底物SPT5调控转录进程的机制;发现了转录延伸因子PAF1通过INTAC调控转录暂停和释放的方式;揭示了INTAC复合物的RNA核酸内切酶和磷酸酶两个功能模块在转录调控中扮演了不同的角色;并首次发现INTAC能够通过单链DNA结合模块SOSS参与维持基因组稳定性的调控;此外,发现了INTAC的PP2A磷酸酶与PNUTS-PP1磷酸酶在转录调控中的拮抗功能。本项研究的发现,进一步完善了对INTAC辅助模块功能的了解,对INTAC各功能模块之间的差异调控有了更深刻的认识,并进一步深入理解。
复旦大学生物医学研究院和同济大学医学院联合培养19级直博生樊鹏宇,复旦大学生物医学研究院博士后尚雪莹、宋爱霞,中国科学院分子细胞科学卓越创新中心博士生陈硕为本研究共同第一作者。复旦大学生物医学研究院/附属肿瘤医院陈飞研究员、中国科学院分子细胞科学卓越创新中心/杭州高等研究院姜海研究员和同济大学附属上海市肺科医院张鹏教授为共同通讯作者。
原文链接:
https://www.nature.com/articles/s41589-024-01807-x
1 Chen, F. X., Smith, E. R. & Shilatifard, A. Born to run: control of transcription elongation by RNA polymerase II. Nat Rev Mol Cell Biol19, 464-478, doi:10.1038/s41580-018-0010-5 (2018).
2Rathert, P. et al. Transcriptional plasticity promotes primary and acquired resistance to BET inhibition. Nature 525, 543-547, doi:10.1038/nature14898 (2015).
3 Zheng, H. et al. Identification of Integrator-PP2A complex (INTAC), an RNA polymerase II phosphatase. Science 370, eabb5872, doi:10.1126/science.abb5872 (2020).
4 Hu, S. et al. INTAC endonuclease and phosphatase modules differentially regulate transcription by RNA polymerase II. Mol Cell 83, 1588-1604 e1585, doi:10.1016/j.molcel.2023.03.022 (2023).
5 Sabath, K. et al. INTS10-INTS13-INTS14 form a functional module of Integrator that binds nucleic acids and the cleavage module. Nat Commun 11, 3422, doi:10.1038/s41467-020-17232-2 (2020).
6 Xu, C. et al. R-loop-dependent promoter-proximal termination ensures genome stability. Nature 621, 610-619, doi:10.1038/s41586-023-06515-5 (2023).