Science丨徐彦辉团队揭示转录起始连续动态全过程
真核细胞的基因转录需要经历起始,延伸,终止等多个阶段。多细胞生物为了满足在同一套基因组的基础上实现差异性的基因表达,需要经过非常复杂且精细的基因表达调控过程。基因表达调控主要集中发生在转录的起始阶段,细胞中基因的转录默认是低活性状态,需要经历一系列复杂的转录起始过程,才能起始基因表达。RNA聚合酶II(Pol II)本身不具备结合和打开基因启动子的能力,需要与6个通用转录因子(general transcription factors,GTFs)包括TFIID、TFIIA、TFIIB、TFIIF、TFIIE以及TFIIH共同在启动子上组装形成转录前起始复合物(preinitiation complex,PIC),才能起始转录。在ATP的驱动下,PIC打开转录起始位点(transcription start site,TSS)附近的DNA双链,DNA模板链进入Pol II活性中心,以NTP为素材,开始生成RNA。转录起始过程涉及十余个复合物上百个蛋白的巨大转变,过去几十年来,众多的课题组利用生物化学、单分子生物物理以及结构生物学等方法开展了大量探索性的工作,但对其发生过程和分子机制的理解还远远不够深入。
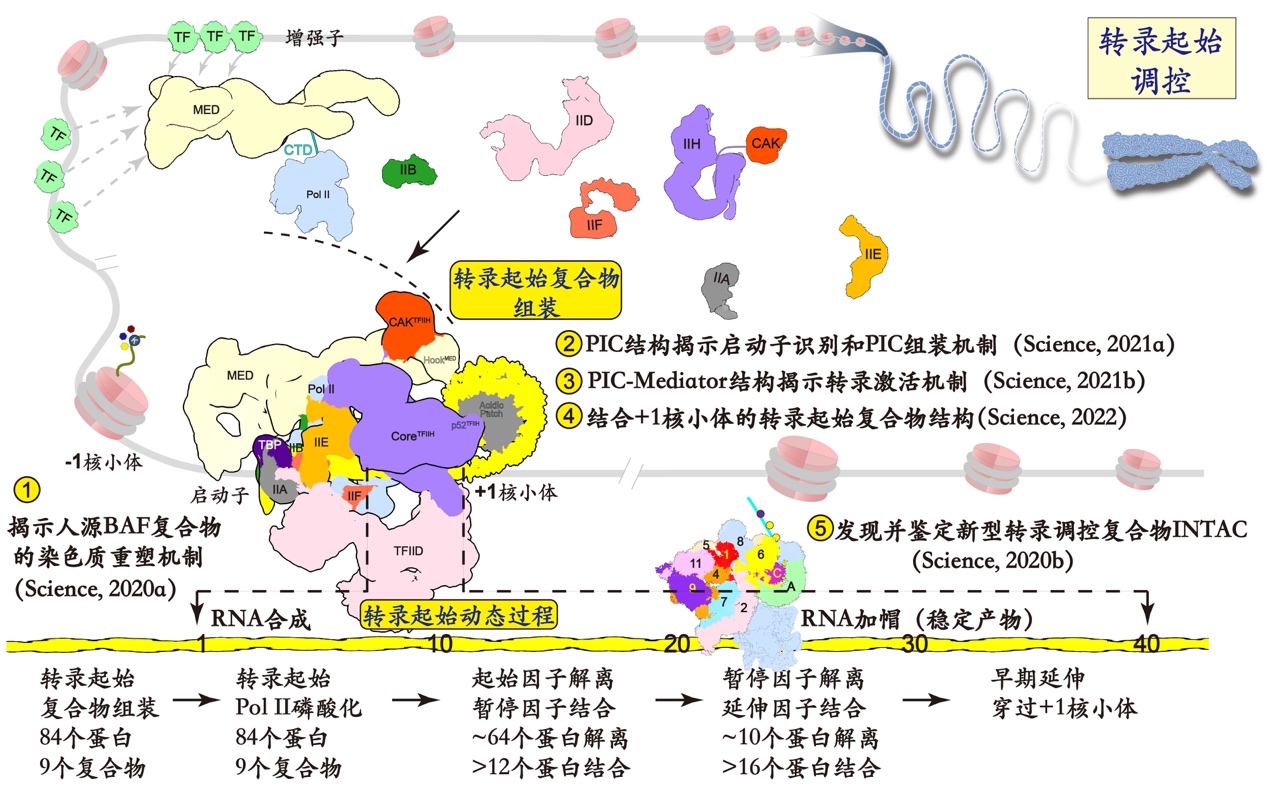
图1.转录起始调控示意图
近几年,我院徐彦辉团队连续在Science杂志发表5篇研究论文,揭示多个转录起始关键过程的分子机制,包括揭示人源BAF复合物的染色质重塑机制、揭示转录起始复合物识别基因启动子及其动态装配机制、揭示中介体促进RNA聚合酶磷酸化和转录激活机制、揭示+1核小体调控转录起始的分子机制以及发现并鉴定新型转录调控复合物INTAC【1-5】 。上述工作较为系统的揭示了转录起始各个阶段关键点的复合物状态,推动了对转录机制的深入理解。 但上述工作,以及目前几乎所有的研究工作,都没有展示出转录起始从双链DNA打开,RNA合成,到聚合酶在DNA上运动这些动态过程,我们对转录的理解仍然停留在静止的阶段。
过去数十年积累的大量生化研究提出了转录起始阶段会发生显著的功能和蛋白组成的变化。当转录开始时,TFIIH将转录起始位点附近的DNA双链打开,模板链进入Pol II的活性中心,PIC转变形成了初始转录复合物(initially transcribing complex, ITC);随后,Pol II以NTP为底物,催化合成RNA,期间经历一系列复杂的结构和复合物组成变化,ITC转变形成早期延伸复合物(early elongation complex, EEC),进入后续的延伸过程。这些生化实验描绘出了转录起始的大致过程,但由于实验设计和实验材料的不一致,不同的实验结论之间相互矛盾,许多结论长期以来未得到统一。尽管领域内进行了几十年的生化和结构研究,但对于转录从头起始如何发生、ITC如何转变成ECC、转录泡为何崩塌,以及转录起始早期过程中潜在的分子机制仍然存在很多问题未被解答。
2023年12月22日,徐彦辉团队在Science杂志上在线发表题为”Structural visualization of transcription initiation in action”的研究长文(Research Article)。该项研究首次用结构重现出了转录从头起始的连续动态全过程,揭示了通用转录因子(GTFs)和转录泡协同RNA聚合酶Pol II调控转录起始向转录延伸转变的分子机制。

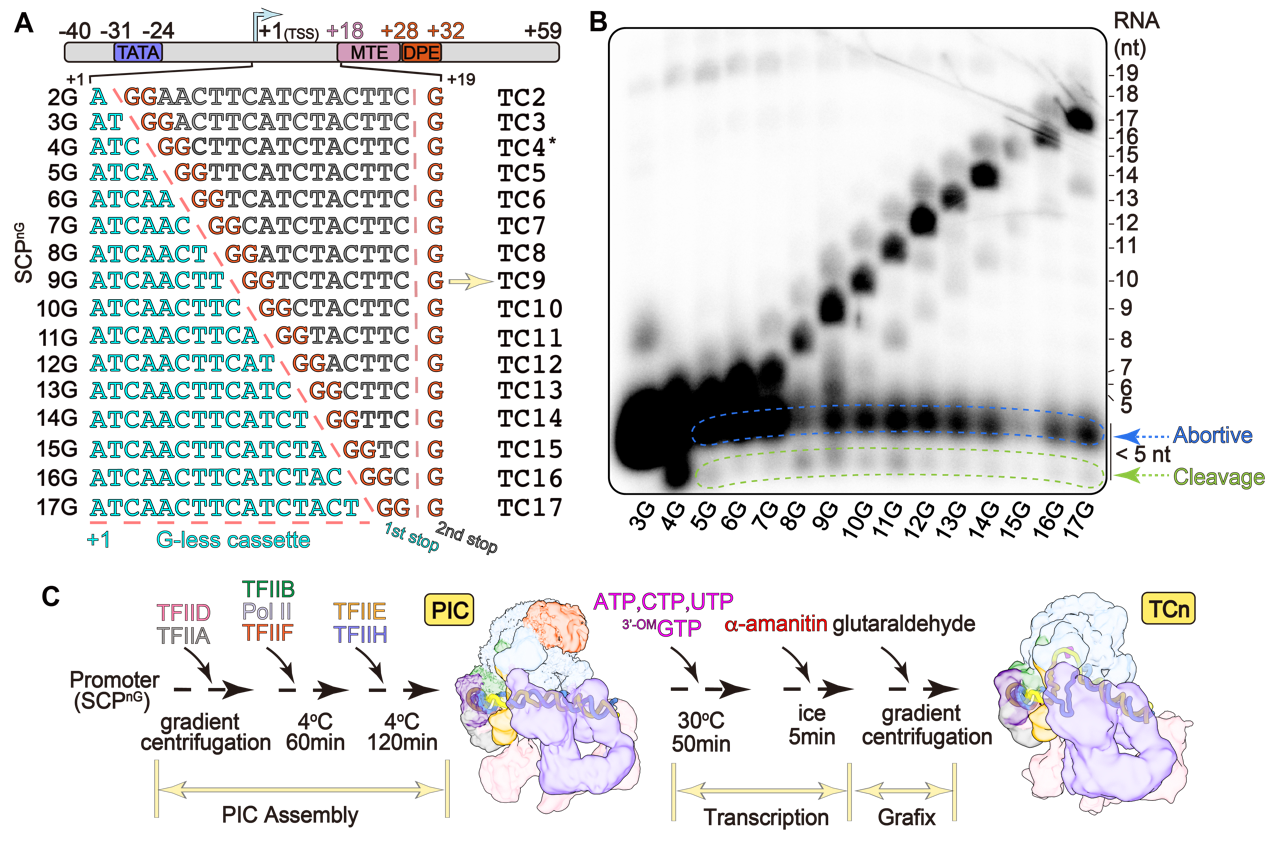
图2.体外重建转录复合物体系。(A)16个(分别暂停在转录起始位点下游2-17位核苷酸的位置)的G-less DNA模板示意图。(B)体外转录活性实验。(C)转录复合物体外组装示意图。
研究团队通过在G-less DNA模板(从转录起始位点TSS到转录暂停位点中间的非模板链不包含G,转录暂停位点处添加GG)上组装转录前起始复合物PIC,随后通过加入ATP, CTP, UTP以及GTP类似物(3’-O-methly-GTP)起始转录反应(图2)。利用该转录体系,研究人员将转录反应人为的暂停在转录起始位点下游2-17位核苷酸的位置,随后结合体外活性实验分析以及冷冻电镜单颗粒技术展开研究。
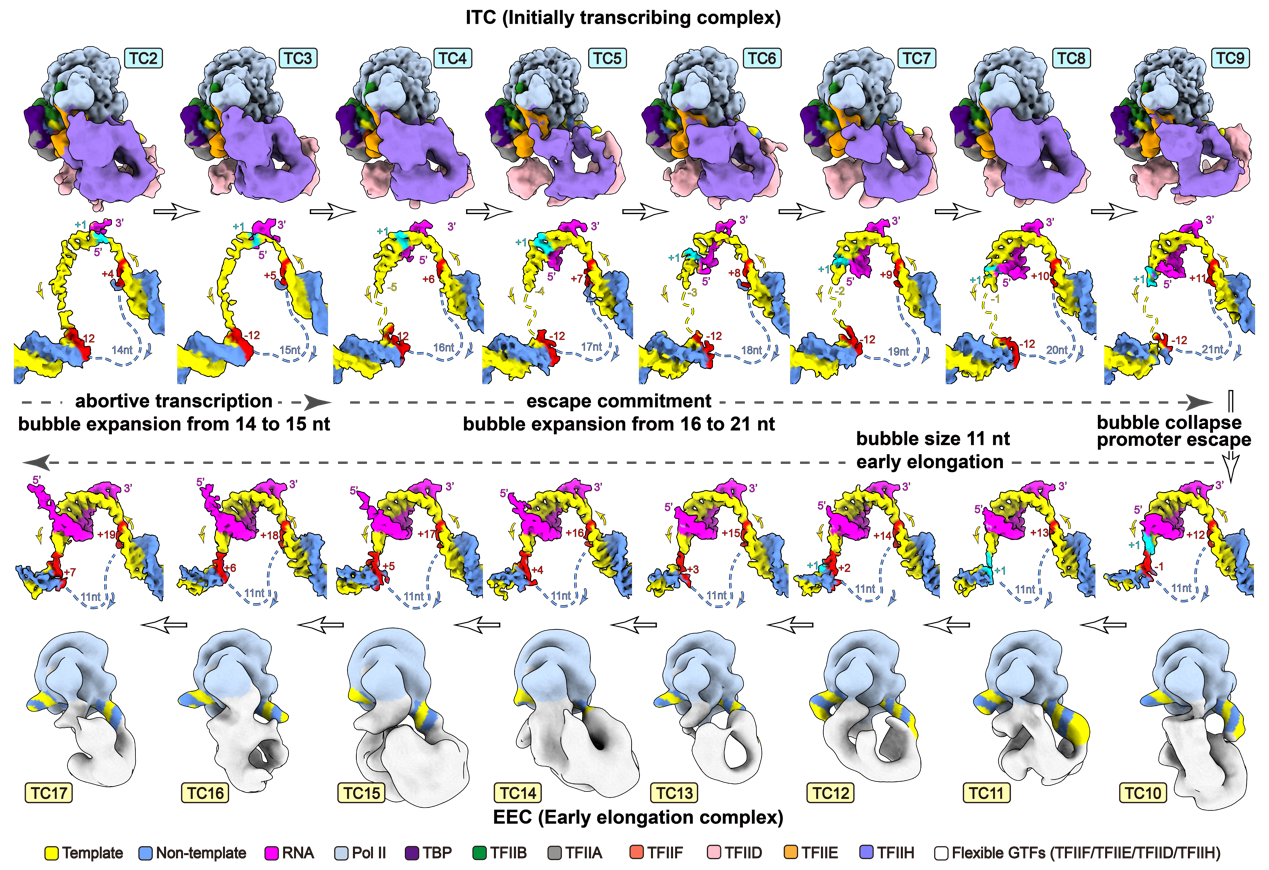
图3. 解析的16个转录复合物结构。
由于复合物的组成和构象不均一,为了获得高分辨率结构数据,研究人员利用300 kV冷冻电镜上采集了约110天,共580,409张照片数据,获得了分别暂停在转录起始位点下游2-17位核苷酸,核心分辨率为2.7-3.3 Å,共16个转录复合物(TC2-TC17)的结构(图3)。通过这16个连续的结构,结合早期该课题组解析的转录前起始复合物PIC结构,研究团队描绘出了连续的转录起始动态全过程,揭示完整转录起始过程及其分子机制(视频1)。
视频1. Pol II转录起始过程
转录开始时,PIC中的TFIIH利用ATP水解产生的能量,使下游启动子移位(translocase),DNA双链打开,模板链进入Pol II的催化中心,形成一个包含约13个nt的转录泡(transcription bubble)的开放复合物(OC/TC1)。随着NTP的加入,OC转变为初始转录复合物ITC(TC2到TC9),RNA开始合成并逐步经历由流产转录(abortive initiation)向逃逸决定(escape commitment)状态的转变,解开的模板单链引导新生RNA合成,随着RNA链从2到9个nt的逐步延长,RNA与模板链的结合逐步紧密。在此过程中,Pol II与启动子上游以及GTFs保持紧密结合,转录泡从14个nt逐步扩大到21个nt。
当RNA链由9nt增加到10nt时,NTP水解驱动RNA-DNA移位并推动所累积的模板单链胀破由TFIIB和Pol II形成的模板链通道,导致TFIIB连带其他GTF从Pol II上解离,转录泡“崩塌”(bubble collapse),转录复合物由ITC转变成EEC状态,Pol II从启动子逃逸(promoter escape)。逃离起始区域的Pol II以EEC的状态(TC10-TC17)继续在DNA上转录。这一复合物后续会发生转录暂停、RNA盖帽、暂停释放、稳定延伸、终止等过程(机制待未来揭示)。
为什么起始过程中转录复合物需要经历如此大的转变?
为了避免错误转录的发生,高等生物进化出复杂的转录起始复合物,利用GTFs帮助Pol II结合和打开基因启动子,因此GTFs对于正确的转录起始是极其必要的存在。但由于GTFs、Pol II和启动子之间的稳定相互作用也会对Pol II离开起始区域造成了巨大的阻碍。为了保证Pol II能够成功的逃离起始区域,进入转录延伸阶段,这些GTFs、Pol II和启动子之间的相互作用必须被破坏。
在早期ITC(early ITC, TC2-TC3)中,Pol II活性中心和模板链通道可以容纳模板链,允许转录泡扩张和初始不稳定的短RNA的合成。在晚期ITC(late ITC, TC4-TC9)中,随着RNA的合成,RNA-DNA杂交链逐渐稳定,二者稳定的结合为支持NTP所驱动的DNA-RNA移位以及GTFs-Pol II-启动子之间相互作用的瓦解提供了基础。在最终形成的EEC中,结合松散的GTFs不再限制Pol II停留在启动子上,使得Pol II能够轻松逃离起始区域,进入下一延伸阶段。因此,GTFs和转录泡的协同变化完美的支持了Pol II从需要GTFs结合并打开启动子进行初始RNA合成到后续Pol II需要GTF解离和转录泡崩塌以逃离启动子进入稳定RNA生产的功能性过渡。
这项工作研究将过去几十年来数百篇关于转录起始研究中的众多看似矛盾的结论作了统一,这些矛盾结论的产生可能是因为实验条件局限性和体系差异造成。工作回答了起始过程中众多问题,揭示了转录起始调控的机制,为理解基因表达调控奠定了结构基础。该项工作也开创了利用生化和结构生物学重构生物大分子机器动态过程,揭示其工作机制的新路径,为众多复杂过程的研究提供了新思路。
复旦大学生命科学学院青年研究员陈曦子、复旦大学生物医学研究院2019级直博生刘维达、青年研究员王茜敏以及粤港澳大湾区精准医学研究院博士后王鑫鑫为本文共同第一作者,复旦大学附属肿瘤医院/生物医学研究院双聘研究员徐彦辉为通讯作者。
原文链接:https://doi.org/10.1126/science.adi5120
参考文献:
1.Chen, X., Wang, X., Liu, W., Ren, Y., Qu, X., Li, J., Yin, X., and Xu, Y.* (2022) Structures of +1 nucleosome-bound PIC-Mediator complex. Science 378, 62-68
2.Chen, X., Yin, X., Li, J., Wu, Z., Qi, Y., Wang, X., Liu, W., and Xu, Y.* (2021) Structures of the human Mediator and Mediator-bound preinitiation complex. Science 372, eabg0635
3.Chen, X., Qi, Y., Wu, Z., Wang, X., Li, J., Zhao, D., Hou, H., Li, Y., Yu, Z., Liu, W., Wang, M., Ren, Y., Li, Z., Yang, H., and Xu, Y.* (2021) Structural insights into preinitiation complex assembly on core promoters. Science 372, eaba8490
4.Zheng, H., Qi, Y., Hu, S., Cao, X., Xu, C., Yin, Z., Chen, X., Li, Y., Liu, W., Li, J., Wang, J., Wei, G., Liang, K., Chen, F. X.*, and Xu, Y.* (2020) Identification of Integrator-PP2A complex (INTAC), an RNA polymerase II phosphatase. Science 370, eabb5872
5.He, S., Wu, Z., Tian, Y., Yu, Z., Yu, J., Wang, X., Li, J., Liu, B., and Xu, Y.* (2020) Structure of nucleosome-bound human BAF complex. Science 367, 875-881