Nature Communications丨屈前辉课题组与合作者报道糖基磷脂酰肌醇转酰胺酶复合物的结构与底物识别机制
糖基磷脂酰肌醇(glycosylphosphatidylinositol,GPI)修饰是真核生物中普遍存在的翻译后修饰。许多可溶蛋白通过GPI修饰而锚定在细胞膜上,行使信号转导、催化、细胞黏附等,影响受精、神经发育和免疫等生物学功能。糖基磷脂酰肌醇转酰胺酶(GPI transamidase,GPI-T)是催化GPI锚定蛋白生物合成途径中的关键酶,主要包含PIGK、PIGS、PIGT、PIGU及GPAA1五个亚基,其成员的基因突变与多种疾病相关。GPI-T酶复合物负责将待修饰的前体蛋白羧基C末端的信号肽切除,并将糖脂分子GPI连接到新暴露的C末端。不同于大多数蛋白水解酶(如TEV酶)选择性酶切特定肽段,GPI-T识别的底物肽段序列不具有唯一性,其切割位点(w位点)通常为侧链较小的氨基酸(Glycine/Alanine),且切割位点至C末端疏水段由~10个亲水氨基酸残基的肽段相连(图1a)。
2022年5月12日,我院屈前辉课题组与中科院分子细胞卓越创新中心(上海生化与细胞所)的李典范课题组、四川大学生物治疗国家重点实验室苏昭铭团队合作在Nature Communications期刊上,在线发表了题为“Molecular insights into biogenesis of glycosylphosphatidylinositol anchor proteins”的研究工作,解析了人源GPI-T酶复合物的高分辨率冷冻电镜结构,结合大量生化实验,分析探讨了GPI-T识别底物GPI糖脂分子的作用机制。

本研究解析了人源GPI-T复合物的2.53 Å高分辨率冷冻电镜三维结构,揭示了其异源五聚体(各1个亚基)的组装机制(图1b)。结构分析、突变实验和活性分析结果提示GPI-T采用与Legumain/Caspase类水解酶类似的底物识别与剪切机制(图1c)。在此结构中还发现了一个内源性GPI糖脂分子被多个亚基共同构建的“组合型”口袋结合(图1d)。此外还有多个脂类分子与GPI-T蛋白紧密作用。通过结构与功能分析,作者们提出了一个模糊底物的识别机制模型。在这个模型中,GPI-T的数个亚基将催化中心固定在一个与膜边界相隔约22 Å的位置(图1d)。这种空间排列符合GPI底物和前体蛋白底物信号肽的膜拓扑关系,从而实现对模糊底物的特异性识别。
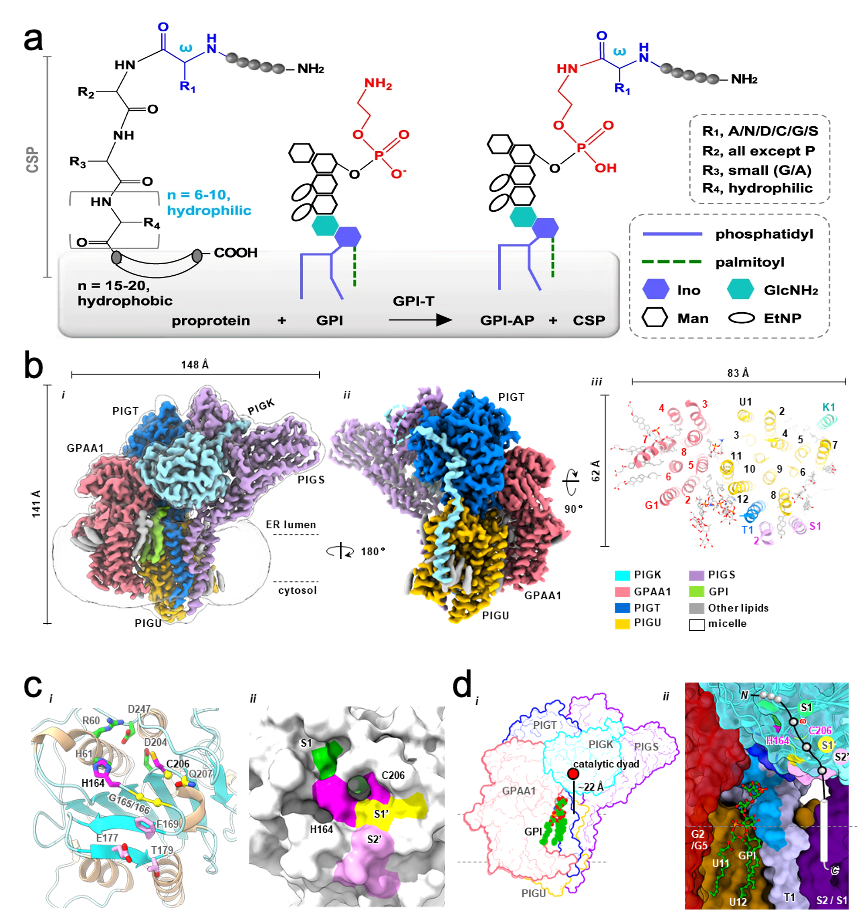
图1. GPI-T的冷冻电镜结构及其活性中心组成。a, GPI-T催化GPI锚定蛋白生物合成示意图。b, GPI-T的冷冻电镜密度图(i,ii)及其跨膜区从内质网腔的俯视图(iii)。不同的亚基及配体使用图中所示颜色标注。c, GPI-T的PIGK亚基的活性中心位点(C206与H164)及可能的底物结合口袋的ribbon(i)及表面图(ii)。其中S1,S1’,S2’分别对应w,w+1,w+2氨基酸残基的可能结合位点。黄色小球表示负氧中心位点。d, GPI结合位点的全景图(i)与局部放大图(ii)。根据此位点信息,亚基PIGK上的催化位点(红色圆点)到膜边界(虚线)的位置约为22埃,此距离与GPI和C端信号肽中的亲水部分长度大致相当。PIGK上的底物结合位点S1/S1’/S2’用b图中颜色标注。前体蛋白用细线加白色C端信号肽表示。N和C分别表示前体蛋白的N和C端。在放大图中,字母加数字表示亚基(G, GPAA1; U, PIGU; T, PIGT; S, PIGS)的第N个跨膜螺旋。
中科院分子细胞科学卓越创新中心博士生徐乙丹、博士后李婷婷,四川大学生物治疗国家重点实验室博士生贾国文,复旦大学生物医学研究院/附属口腔医院博士后周子璇为本文共同第一作者。罗椅天博士,晁钰琳同学和鲍娟博士做了重要贡献。中科院分子细胞科学卓越创新中心研究员李典范、复旦大学生物医学研究院青年研究员屈前辉和四川大学生物治疗国家重点实验室研究员苏昭铭为本文的共同通讯作者。
原文链接:https://www.nature.com/articles/s41467-022-30250-6