Genome Biology丨杨力组开发基于深度学习的计算分析框架实现RNA测序数据直接鉴别RNA编辑与DNA突变位点
2024年10月08日,Genome Biology在线发表了复旦大学生物医学研究院杨力研究组题为“DEMINING: a deep learning model embedded framework to distinguish RNA editing from DNA mutations in RNA sequencing data”的最新研究成果,本研究发布了一套新型计算分析框架——DEMINING,可以从RNA测序数据直接鉴别RNA编辑与DNA突变位点(https://genomebiology.biomedcentral.com/articles/10.1186/s13059-024-03397-2)。

人类转录组中存在大量的RNA突变信息,其主要是人腺苷脱氨酶(ADAR)催化的腺苷(A)到肌苷(I)的RNA编辑(A-to-I RNA editing, REs),如何能从海量转录组数据中排除测序错误、比对错误以及DNA突变等信息的干扰,有效且精准地识别这些RNA编辑位点是转录组计算生物学领域的研究难点。近期,以深度学习为代表的人工智能方法,在各个领域展现出强大的应用前景,为解决如何从RNA测序数据中精确区分RNA编辑与DNA突变的这一难题提供了新的思路。本研究开发的DEMINING流程(图1),通过深度学习模型DeepDDR,实现从RNA测序数据中高效准确地识别RNA编辑和DNA突变。DEMINING流程首先通过严格的筛选标准去除转录组数据中的测序和比对错误(图1a),然后将获得的高可信度(DNA和RNA)突变位点信息作为输入,通过搭建的深度学习模型DeepDDR实现DNA突变和RNA编辑的精准区分(图1b)。在DeepDDR模型的训练过程中,研究团队从403个配套的RNA测序和DNA测序数据集中提取了122,872个高可信度的RNA编辑位点和相同数量的DNA突变位点,分别用于训练、验证和测试模型。在此模型构建过程中,研究人员创新性的将突变位点上下游序列和测序读段编码,构建带注意力的双碱基上下文共轭同频矩阵(matrix of the co-occurrence frequencies of each mutation site with its context bases, CMC),作为DeepDDR模型的编码输入,接着通过使用多个层次的卷积和池化操作,提取出突变位点周围的序列以及读段比对特征,通过这种方法,DeepDDR模型不仅能够识别出突变位点,还能够捕捉到这些突变在更大范围内的上下文信息,这可能是有效区分RNA编辑和DNA突变的关键所在。
DEMINING框架不仅在人类样本中表现出色,经过小样本迁移学习,还可应用于非灵长类的RNA测序样本,展现了DEMINING框架广泛的应用潜力。在迁移学习过程中,研究团队将人类数据集上训练的DeepDDR模型作为预训练模型,进一步利用小鼠脑组织的RNA测序数据进行微调。通过这种方法,DeepDDR模型在小鼠数据集上的RNA编辑识别准确性得到了显著提高。相似的改进也在其他非灵长类物种(线虫)的数据集中得到了验证,表明DEMINING框架的可迁移性和广泛适用性。
最后,科研人员探索了利用DEMINING框架从已发表的人类RNA测序数据中直接鉴别DNA突变的应用。在分析急性髓性白血病(AML)患者的RNA测序数据时,DEMINING识别出了大量此前未报道的可能与疾病相关的DNA突变和RNA编辑位点。这些突变与宿主基因的上调表达或新抗原的产生相关,为AML的发病机制提供了新的见解。
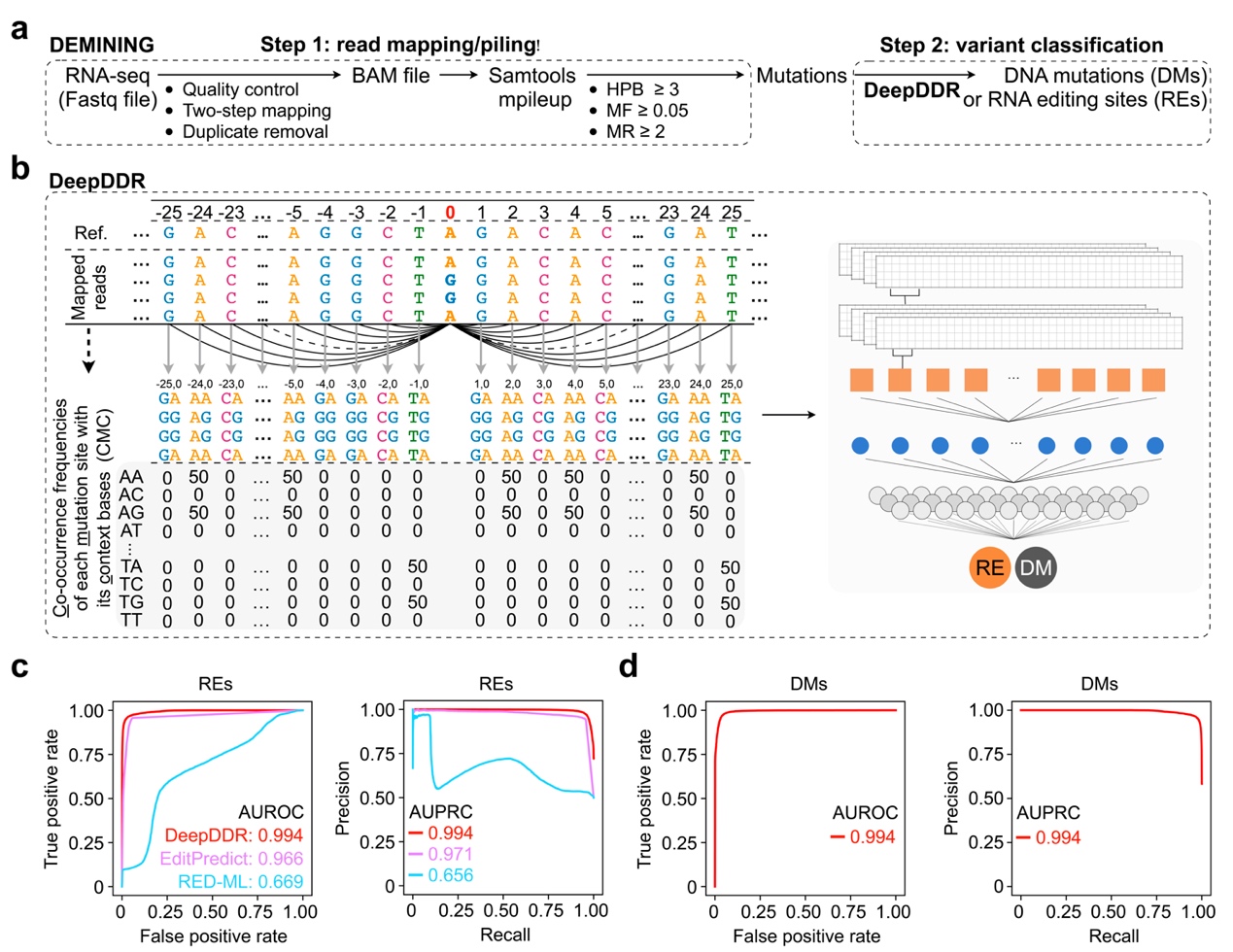
图 1 | 开发用于DNA突变(DMs)和RNA编辑位点(REs)分类的DEMINING框架及嵌入式DeepDDR模型
a. 构建用于直接分类DNA突变(DM)和RNA编辑(RE)的分步DEMINING计算框架。HPB:每十亿碱基上的读段覆盖数,MF:突变频率,MR:包含突变的读段数目。b. 用于DM和RE分类的嵌入式DeepDDR模型示意图。左图:通过每个突变位点与其上下文碱基的共现频率(CMC)提取特征的策略。右图:DeepDDR模型架构。c. 在RE识别上评估不同模型。展示了DeepDDR(红色)、EditPredict(紫色)和RED-ML(蓝色)在测试集上RE识别性能的ROC(左图)曲线和精度-召回率(PRC,右图)曲线。图中标明了三种方法的ROC下面积(AUROC)和PRC下面积(AUPRC)值。 d. 在DM识别上评估DeepDDR。展示了DeepDDR在测试集上DM识别性能的ROC(左图)和PRC(右图)。图中包括了DeepDDR的AUROC和AUPRC值。
综上,DEMINING框架通过嵌入的深度学习模型DeepDDR,实现了从RNA测序数据中高效、精确地鉴定RNA编辑和DNA突变。随着RNA测序数据的不断积累,特别是与疾病相关的转录组数据,DEMINING框架有望在更广泛的人类疾病RNA测序样本中应用,揭示更多与疾病相关的突变和基因,为诊断和治疗提供潜在靶点。DEMINING的构建是利用人工智能模型在转录组分析中的有一有效尝试。2024年10月3日,杨力研究员也受邀与国际同行为Molecular Cell撰写相关Voices (https://www.cell.com/molecular-cell/abstract/S1097-2765(24)00693-2),展望人工智能时代转录组RNA系统分析研究的发展趋势(Yang et al, Molecular Cell 2024)。