Genome Biology丨胡璐璐课题组开发RNA修饰检测新技术,可在极低量的RNA样本中精确检测表观转录组修饰
复旦大学生物医学研究院胡璐璐课题组与同济大学康九红课题组联合开发了一种创新的文库构建策略Uli-epic,该策略结合了化学基础的RNA修饰检测技术,可在极低量的RNA样本中精确检测表观转录组修饰。相关研究成果于2025年11月11日以Uli-epic: profiling RNA modifications from ultra-low input samples为题已发表于Genome Biology杂志。

Uli-epic BID-seq将Uli-epic与BID-seq(1,2)技术相结合,能够在单核苷酸分辨率下精确量化Ψ修饰,并将检测限从10 ng的mRNA降低至100 pg。类似地,Uli-epic GLORI结合了Uli-epic与GLORI(3,4)技术,在单核苷酸分辨率下对m6A修饰进行精确量化,将检测限从200 ng的mRNA降低至10 ng。利用Uli-epic BID-seq与Uli-epic GLORI技术,研究人员成功地在HEK 293T细胞、野生型(WT)和胎儿生长受限(FGR)小鼠的神经干细胞(NSCs)以及精子中, 对极少量的RNA样本进行了Ψ和m6A修饰位点的分析。
研究表明,在FGR NSCs和精子中,与WT相比,一些mRNA和tRNA的Ψ修饰丰度存在显著差异。已知mRNA中的Ψ修饰能够影响核糖体对tRNA的选择,进而增加人体细胞中的氨基酸置换水平。因此,进一步研究FGR小鼠NSCs和精子的翻译效率及氨基酸序列的变异性,成为值得深入探讨的方向。特别是,tRNA-Arg-CCT-4-1中的Ψ27/Ψ28和tRNA-Arg-ACG-3-1中的Ψ54/Ψ55在FGR NSCs中的丰度上调,而tRNA-Leu-TAG-3-1和tRNA-Arg-CCT-4-1中Ψ54/Ψ55的丰度在FGR精子中显著降低。此外,FGR精子中的Ψ54/Ψ55丰度相较于WT精子明显下降。特别有趣的是,tRNA-Arg-CCT-4-1中的Ψ54/Ψ55在FGR NSCs中上调,但在FGR精子中下调。
已有研究表明,人类PUS10作为假尿苷合成酶,催化了tRNA TΨC臂中Ψ54和Ψ55的合成。小鼠的假尿苷合成酶1(Pus1p)已被证明能够催化体外合成的tRNA中Ψ27、Ψ28、Ψ34和Ψ36位置的假尿苷形成。因此,研究PUS10和Pus1p在FGR小鼠NSCs及生殖细胞中的表达差异及功能作用,具有重要的意义。此外,探究tRNA TΨC臂与核糖体分子相互作用的机制,特别是受Ψ54/Ψ55丰度变化调控的机制,以及Ψ27/Ψ28丰度变化对覆盖片折叠的影响,值得进一步研究。
与假尿苷修饰的研究相对较少相比,m6A修饰在生殖系统和神经系统中发挥着至关重要的作用,并且作为关键调控因子参与代际遗传。研究人员在FGR NSCs和精子中发现了41个共同的差异性m6A修饰位点。功能富集分析显示,这些位点的相关基因与转录调控和突触功能密切相关,提示它们可能在FGR相关的子代认知障碍及代际传递机制中发挥作用。
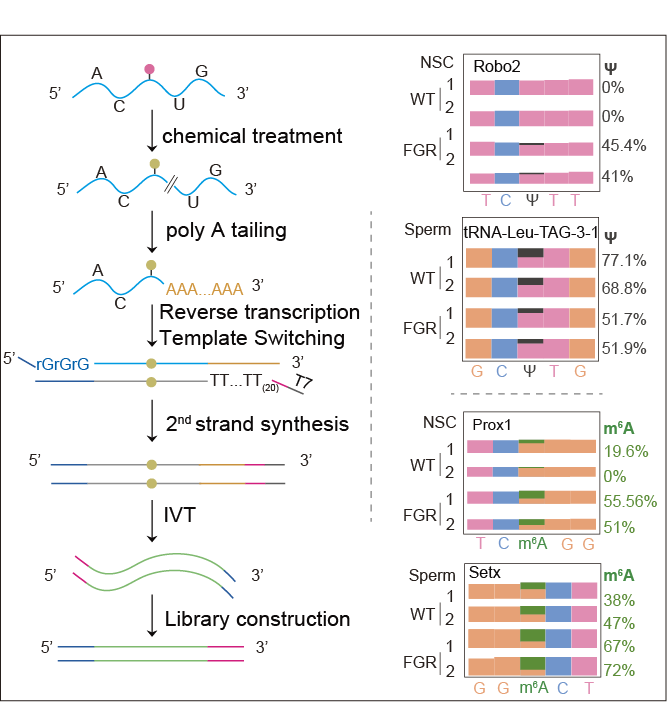
图1:胡璐璐课题组研发的Uli-epic技术流程(左)与使用Uli-epic检测到的胎儿生长受限(FGR)小鼠的神经干细胞(NSC)和精子(sperm)的异常m6A和假尿嘧啶(Ψ修饰)。
综上所述,这些发现表明,针对假尿苷合成酶(如PUS10、Pus1p)以及与NSCs和精子中共同m6A修饰相关的基因/通路的靶向干预,可能为改善FGR子代认知障碍等不良神经发育结局并阻断其代际传递提供潜在的治疗策略。
胡璐璐课题组此前已开发了LAMP-MS方法(5),通过质谱阵列分析可视化量化RNA的m6A修饰。本研究引入的Uli-epic策略,结合LAMP-MS方法,不仅能实现对特定m6A和Ψ修饰位点的可视化量化,且适用于极低起始RNA样本。此策略的结合大大拓宽了液体活检的应用范围,为临床诊断中RNA修饰的应用提供了新的视角。该工作为超低量RNA表观转录组分析提供了有价值的文库构建策略,丰富了表观遗传学的工具箱,并为研究Ψ和m6A修饰在胎儿生长受限中的生物学功能提供了重要数据资源。
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-025-03857-3
1.Dai, Q. et al. Quantitative sequencing using BID-seq uncovers abundant pseudouridines in mammalian mRNA at base resolution. Nat Biotechnol41, 344-354, doi:10.1038/s41587-022-01505-w (2023).
2.Zhang, L. S. et al. BID-seq for transcriptome-wide quantitative sequencing of mRNA pseudouridine at base resolution. Nat Protoc19, 517-538, doi:10.1038/s41596-023-00917-5 (2024).
3.Liu, C. et al. Absolute quantification of single-base m6A methylation in the mammalian transcriptome using GLORI. Nat Biotechnol41, 355-366, doi:10.1038/s41587-022-01487-9 (2023).
4.Shen, W. et al. GLORI for absolute quantification of transcriptome-wide m6A at single-base resolution. Nat Protoc19, 1252-1287, doi:10.1038/s41596-023-00937-1 (2024).
5.Xie, R. et al. LAMP-MS for Locus-Specific Visual Quantification of DNA 5mC and RNA m6A Using Ultra-Low Input. Angew Chem Int Ed Engl64, e202413872, doi:10.1002/anie.202413872 (2025).