Nature Communications丨胡晋川课题组揭示RNA聚合酶II被损伤阻挡后的命运决定机制及其与转录偶联修复缺陷相关遗传疾病的关系
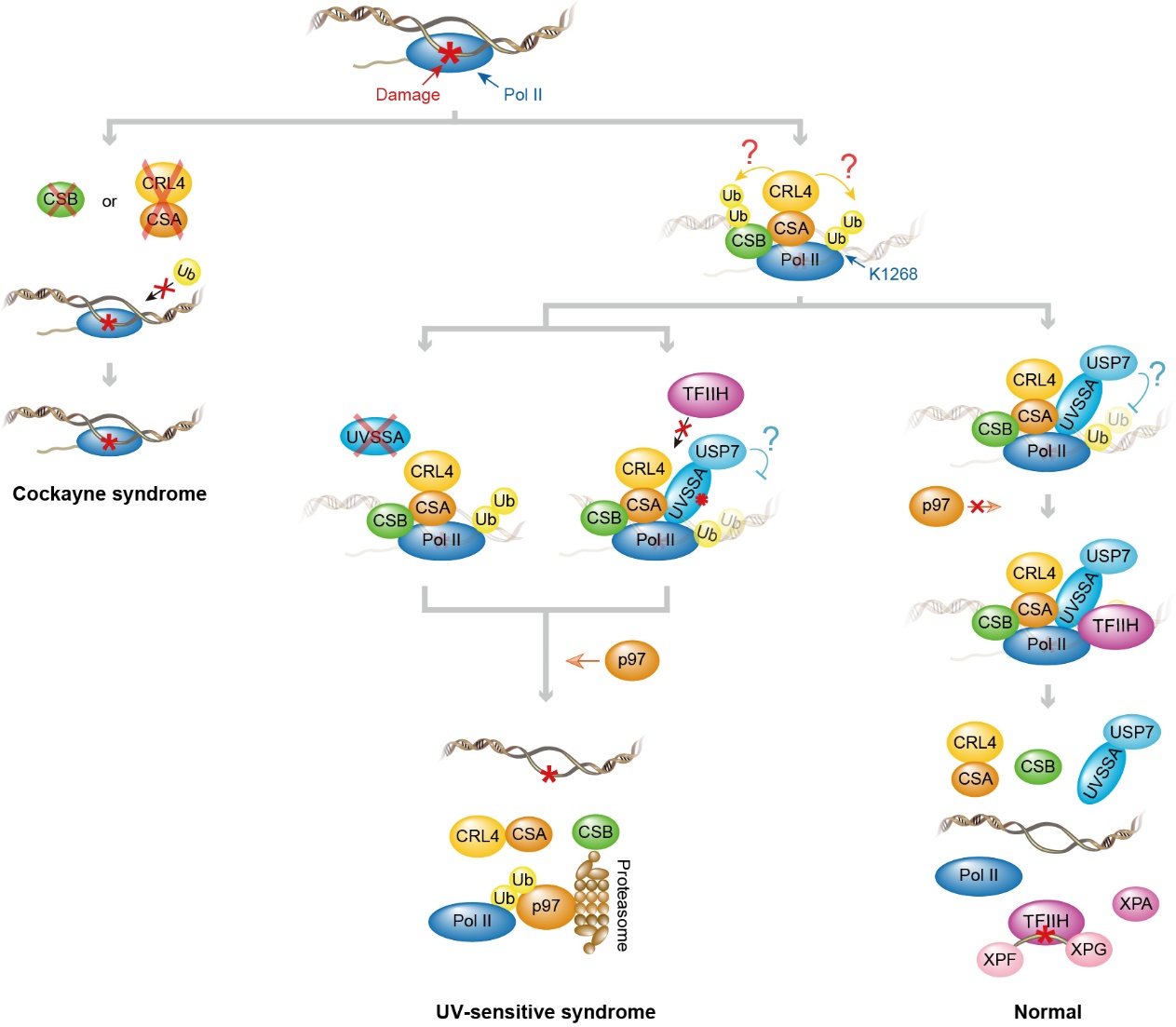
高效准确的转录对生命活动至关重要,多种内源或外源的DNA损伤都会阻挡RNA聚合酶,从而阻碍转录的进行。转录偶联修复(Transcription-Coupled Repair, TCR)可以高效地移除阻挡RNA聚合酶II(PolII)的DNA加合物损伤,恢复正常的转录。转录偶联修复是核苷酸切除修复的亚途径,当PolII被损伤阻挡后,CSB、CSA与UVSSA等转录偶联修复因子会被依次募集。其中CSA所在的CRL4CSA泛素E3连接酶会泛素化CSB、PolII大亚基RPB1以及UVSSA,而UVSSA与USP7形成复合物,通过去泛素化来稳定这些修复因子。随即这些蛋白共同招募核苷酸切除修复途径的下游通用修复因子TFIIH、XPA、RPA以及两个核酸内切酶XPG和XPF,切除损伤所在的一小段单链DNA,最后由DNA聚合酶和连接酶补平缺口,完成修复。
转录偶联修复缺陷会导致不同的遗传疾病,其中CSB和CSA基因缺陷会引起科凯恩综合征(Cockayne
syndrome,CS),患者表现为严重的神经系统发育障碍和早衰,平均寿命仅为12岁;而UVSSA基因缺陷会导致紫外线敏感综合征(UV-sensitive Syndrome,UVSS),患者仅表现为对紫外线敏感,但患皮肤癌风险没有增加,也没有影响神经系统。这几个基因的缺陷都会导致转录偶联修复的完全失活,因此修复本身不是引起不同症状的直接原因。有一种假说认为当修复受阻时被损伤阻挡的PolII是否滞留在损伤位点可能是导致不同表型的原因,因为停滞的PolII可能对复制等过程造成更大的干扰,也会阻碍其它修复机制来处理损伤。然而,由于缺乏直接检测PolII与损伤结合的技术,对修复受阻时被损伤阻挡的PolII的命运目前所知甚少。
2024年8月17日,复旦大学生物医学研究院胡晋川课题组在Nature Communications上发表了题为Coordination of transcription-coupled repair and repair-independent release of lesion-stalled RNA polymerase II的研究论文,发现在转录偶联修复失活时,停滞在损伤位点的PolII以依赖CRL4CSA介导的泛素化的方式,被p97-蛋白酶体途径从损伤位点移除,揭示了导致不同症状的修复基因缺陷对PolII命运的不同影响。

在前期工作中作者开发了能以单碱基分辨率检测蛋白与DNA加合物损伤相互作用的基因组分布的测序方法PADD-seq,发现在转录偶联修复正常细胞中PolII与损伤的结合会在UV照射后2小时内消失,而在修复缺陷的CSB敲除细胞中PolII会长期滞留在损伤位点【1】。在此基础上,他们发现CSA敲除细胞中PolII同样会滞留在损伤位点,而UVSSA敲除细胞则与修复正常细胞类似,PolII从损伤位点快速离开。进一步研究发现,在UVSSA敲除细胞中,PolII的快速解离是由p97-蛋白酶体途径介导,且依赖于CRL4CSA的泛素连接酶活性。然而,p97不参与转录偶联修复,在修复正常的细胞中也不参与移除被损伤阻挡的PolII,提示细胞会优先选择转录偶联修复而不是直接移除被损伤阻挡的PolII。RPB1-K1268位点是UV诱导的主要泛素化位点,其突变会显著抑制转录偶联修复以及PolII的直接解离,提示该位点的泛素化在这两条通路中都扮演了重要角色。最后,UVSSA-USP7的去泛素化活性对转录偶联修复有一定影响,但是不会阻碍p97介导的PolII直接解离。这项工作全面揭示了转录偶联修复受阻时被损伤阻挡的PolII命运决定机制,并为探索转录偶联修复基因相关遗传疾病的分子机制提供了重要线索。
复旦大学生物医学研究院/附属上海市第五人民医院的双聘PI胡晋川是论文的通讯作者,复旦大学生物医学研究院博士研究生祝永昌(已毕业)、张希平和高孟为论文的共同第一作者。本研究获得了科技部重点研发计划和国家自然科学基金面上项目等资助。
值得注意的是,今年7月荷兰Erasmus大学Jurgen A. Marteijn课题组在Nucleic Acids Research杂志上发表了相似的结果【2】,荷兰Leiden大学Martijn S. Luijsterburg课题组也于近期在预印本平台BioRxiv发布了类似的工作【3】。
原文链接:https://www.nature.com/articles/s41467-024-51463-x
参考文献:
1.Zhu, Y. et al. Genome-wide mapping of protein-DNA damage interaction by PADD-seq. Nucleic Acids Res 51, e32 (2023).
2.Gonzalo-Hansen, C. et al. Differential processing of RNA polymerase II at DNA damage correlates with transcription-coupled repair syndrome severity. Nucleic Acids Res (2024).
3.van der Meer, P.J., Yakoub, G., Nakazawa, Y., Ogi, T. & Luijsterburg, M.S. Clearance of DNA damage-arrested RNAPII is selectively impaired in Cockayne syndrome cells. bioRxiv, 2024.05.17.594644 (2024).