Nucleic Acids Res | 杨力团队发布环形RNA大百科全书数据库(CIRCpedia v3)助力环形RNA探索研究
2025年10月22日,复旦大学生物医学研究院/复旦大学附属儿科医院杨力研究组在Nucleic Acids Research杂志在线发表了题为CIRCpedia v3: an interactive database for circular RNA characterization and functional exploration的论文,报道了升级版的环形RNA数据库CIRCpedia(https://bits.fudan.edu.cn/circpediav3)。

反向剪接产生的环形RNA(circRNA)因其特殊的共价闭合环结构,相较于同源线性RNA展现出独特的高级结构特征及低细胞免疫原性。通过多样化的分子机制,环形RNA可广泛参与调控各类细胞活动,并与不同的生理调控和病理状态密切相关。近期,迅速积累的高通量(二代/三代)测序数据揭示了环形RNA的普遍存在,为相关研究提供了宝贵的数据资源。最新报道的CIRCpedia v3数据库系统性整合了来自20个物种的2413个RNA测序样本,覆盖了203种组织类型和375种细胞系以及多种亚细胞组分,成功注释了超过260万个环形RNA。升级版的CIRCpedia v3:1)依据领域内共识,为每个环形RNA提供了规范化命名,准确区分来源于同一基因的多个不同环形RNA,为学术研究和科研交流提供了便利;2)采用统一的FPB参数对环形RNA及同源线性RNA进行标准化定量分析,实现了两者表达水平的直接对比;3)提供了样本间环形RNA表达水平的在线比较分析及可视化展示功能,可以有效协助用户筛选特定环境下潜在的功能性环形RNA;4)集成了多种工具,为环形RNA功能研究提供了全方位的注释信息、功能预测、实验设计和序列优化方案,包括碱基编辑器sgRNA/siRNA/引物设计工具、DIS3偏好降解序列检测及稳定性优化、亚细胞定位预测、RNA/RBP分子相互作用预测分析、m6A修饰位点识别、翻译活性预测等(图1)。
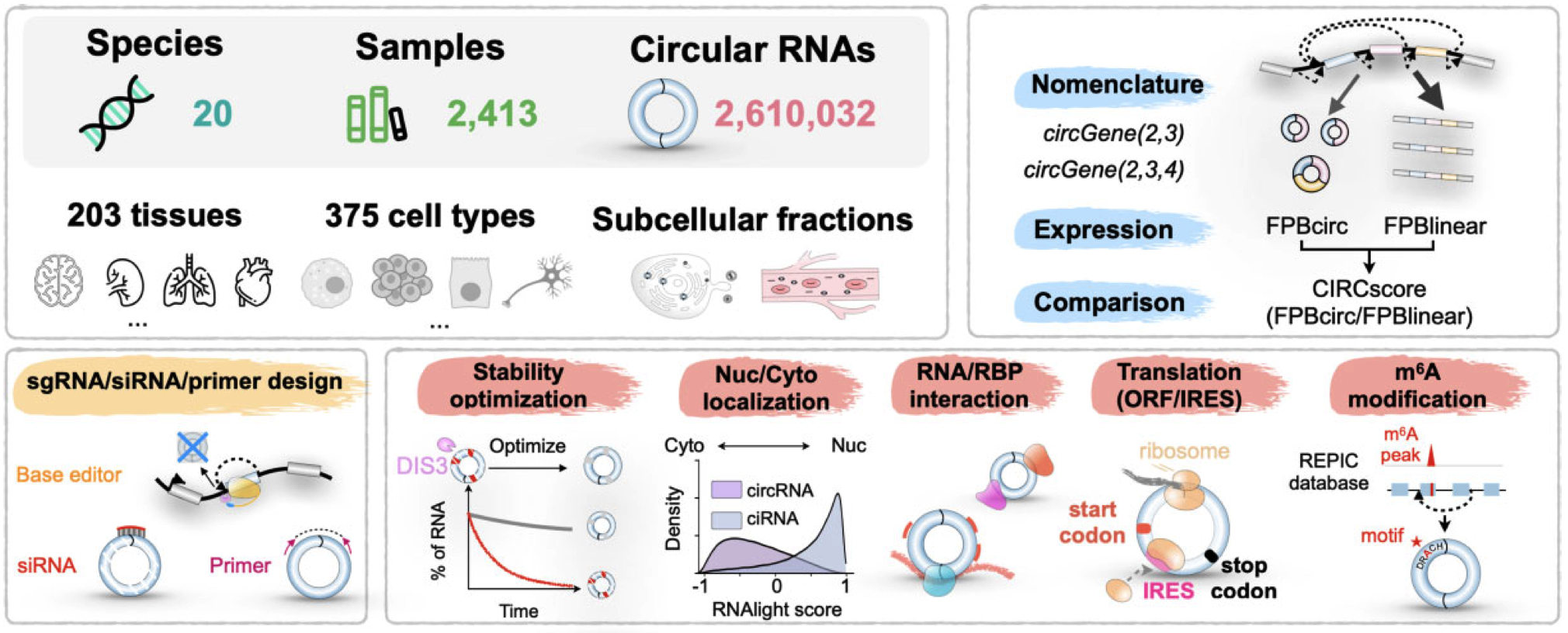
图1 CIRCpedia v3数据库功能概览
综上,升级版的CIRCpedia v3为环形RNA研究提供了涵盖表达鉴定、功能预测到实验验证设计的一站式综合数据资源平台,为环形RNA领域的深入研究提供了有力的数据基础和技术预测支撑。
中国科学院上海营养与健康研究所/复旦大学生物医学研究院联合培养博士研究生翟思楠为本文第一作者,复旦大学生物医学研究院/复旦大学附属儿科医院研究员杨力和青年研究员马旭凯为本文共同通讯作者。该研究得到了中国科学院分子细胞科学卓越创新中心陈玲玲研究员的大力支持和指导,获得国家重点研发计划、国家自然科学基金、中国科学院青年基础研究项目、上海市科学技术委员会等的项目资助。
原文链接 https://doi.org/10.1093/nar/gkaf1039