复旦大学徐彦辉组解析人源ATR-ATRIP结构揭示DNA损伤应答机制
复旦大学徐彦辉课题与清华大学王宏伟、王佳伟课题组合作,于12月22日在细胞研究《Cell Research》杂志,在线发表了题为“Cryo-EM structure of human ATR-ATRIP complex”的研究论文,报道了人源DNA损伤应答关键复合物ATR-ATRIP冷冻电镜结构。
DNA损伤(DNA damage)是导致基因组不稳定,细胞癌变或凋亡的重要来源。ATR(ataxia-telangiectasia mutated- and Rad3-related)是DNA损伤检查点的关键激酶蛋白,传递损伤信号至效应器的核心蛋白启动修复。因此ATR是抗癌药物设计的重要靶标。ATR的功能研究已经有20余年,但至今都没有人源ATR蛋白的高分辨结构,也限制了人们对ATR发挥功能的理解。
研究人员成功解析了人源DNA损伤检查点核心激酶复合物ATR-ATRIP整体分辨率为4.7 Å,催化核心分辨率为3.9 Å的冷冻电镜结构(下图),并构建了催化核心的原子模型,为ATR-ATRIP复合物的组装和发挥功能的分子机制提供了重要的结构信息。
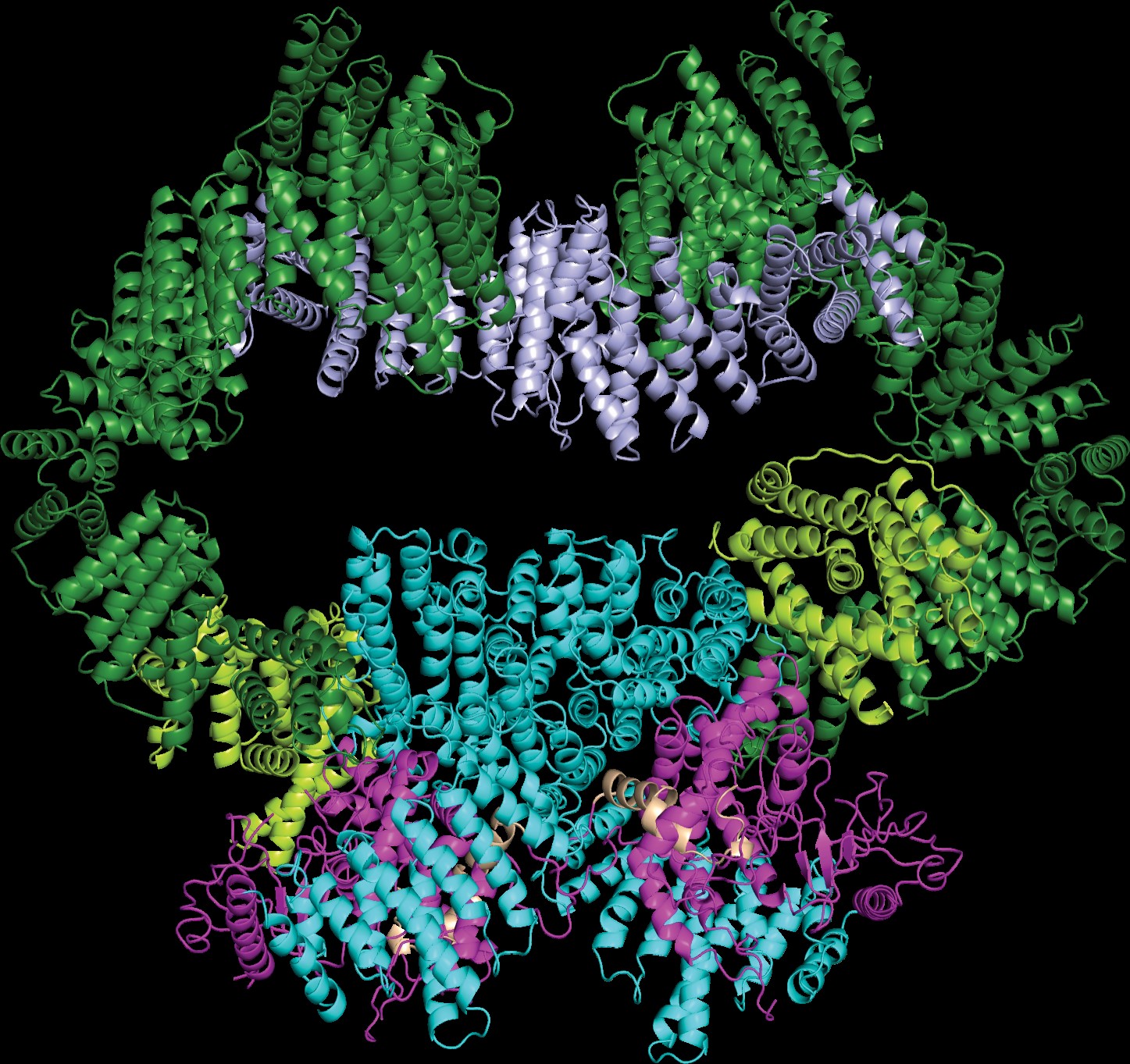
ATR-ATRIP复合物的整体结构
该研究成果阐释了ATR-ATRIP的组装方式与ATR的催化反应的分子机制,发现复合物中ATR形成非对称的二体,其柔性很可能与其功能调控密切相关。根据结构信息搭建的ATR与其异性小分子抑制剂VX-970的模型,为酶活性抑制机理提供了理论依据,为未来抗癌药物设计提供了结构基础。结构分析表明ATR呈现开放的催化中心,不存在抑制结构元件阻挡底物进入催化中心,既底物可以自由进出催化中心并发生磷酸化反应。该构象与人源ATM(ataxia-telangiectasia mutated)的开放构象相似,却与近期蔡刚教授报道的酵母源Mec1(ATR)状态不同。
值得一提的是,ATR与mTOR,DNA-PKcs同属PIKK蛋白激酶家族。徐彦辉课题组前期报了mTORC1复合物和DNA-PK全酶复合物的冷冻电镜结构,上述工作较为系统的阐释了PIKK家族蛋白的共性和特性,为该家族蛋白的结构与功能研究,做出了一定的贡献。
该项工作是复旦大学与清华大学研究人员合作完成,复旦大学博士生饶钦辉和刘梦杰为共同第一作者,徐彦辉、王宏伟、王佳伟为共同通讯作者。徐彦辉为复旦大学附属肿瘤医院研究员,复旦大学生物医学研究院(Institutes of Biomedical Sciences)兼职研究员(课题组网页http://xulab.fudan.edu.cn/index.html)。该项研究工作得到了科技部重点研发计划、国家自然科学基金、中科院先导专项等项目的支持。国家蛋白质设施(上海)、中科院生物物理所和清华大学三家单位的电镜平台在样品制备和数据收集等方面给予了重要的支持