生物医学研究院和化学系的杨芃原团队合作创建了精准N糖蛋白质组学分析方法
近日,复旦大学生物医学研究院和化学系的杨芃原课题组与北京中科院计算所贺思敏课题组和上海蛋白质中心黄超兰团队合作,发表了基于质谱的高通量糖基化肽段分析方法pGlyco2.0,为精准N糖蛋白质组学提供了最新的技术。9月5日,相关研究成果以《pGlyco2.0:基于综合质控和一步质谱法的精准N糖蛋白质组学糖肽分析方法》(pGlyco 2.0 enables precision N-glycoproteomics with comprehensive quality control and one-step mass spectrometry for intact glycopeptide identification)为题在《自然通讯》(Nature Communications)上发表。杨芃原教授、贺思敏研究员和黄超兰研究员为共同通讯作者。杨芃原教授为该文章的Lead Contact。

糖基化是最复杂的蛋白后修饰之一。与其他蛋白后修饰相比,糖基化不但会产生宏观不均一性(每个蛋白上可能有多个后修饰位点),更会产生海量的微观不均一性(每个位点上可能有几十甚至上百种不同的后修饰基团)。除此之外,糖链本身的离子化效率很低。这些因素的结合使得糖基化分析的通量和质量远低于蛋白质组学的常规分析水平。本研究通过深入研究和测试质谱条件,开发基于阶梯能量的一步质谱采集法,提高了糖肽鉴定的通量和开发具有自主产权的pGlyco2.0糖肽检索引擎,从糖链、肽段、糖肽三个层面对糖肽数据库检索进行精确质控,从而大幅提升了N糖蛋白质组学分析的通量和质量。
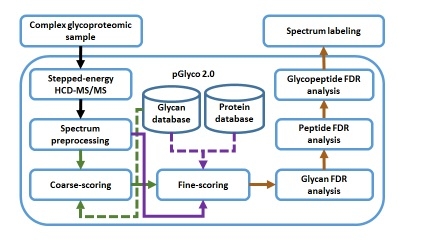
pGlyco2.0流程图
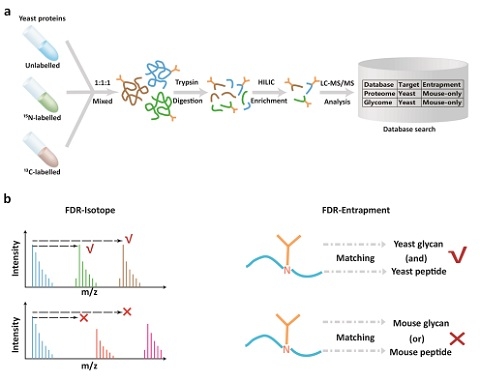
基于重标技术的糖肽质控新方法
此外,该研究首次将重标元素应用于糖肽鉴定准确度分析,为该领域的质控分析提供了新的方法及标准。最后,研究报道了目前最大的糖基化数据集:在1%的假阳性率下,该研究在小鼠的五个脏器中鉴定到了超过一万条N糖肽。