Genomics Proteomics Bioinformatics丨杨力团队构建跨物种单细胞分辨率的多聚腺苷位点大百科全书数据库-PASSpedia
2025年09月23日,复旦大学生物医学研究院/复旦大学附属儿科医院杨力团队在Genomics Proteomics Bioinformatics在线发表了题为PASSpedia: A Polyadenylation Site Database Across Different Species at Single Cell Resolution的论文,报道了一个单细胞分辨率的多物种多聚腺苷酸化位点(polyadenylation sites, PAS)大百科全书数据库-PASSpedia(https://bits.fudan.edu.cn/PASSpedia/)。

真核(mRNA)的成熟依赖于多种共转录及转录后事件的协同调控,包括5'端加帽、(可变)剪接、RNA编辑/修饰以及3'端(多聚腺苷酸化,polyadenylation)加尾等多个关键步骤。多聚腺苷酸化加尾作为一种重要的加工过程,广泛参与RNA命运和功能调控,对于RNA的稳定性、亚细胞定位及翻译效率等至关重要。因此,精确鉴定多聚腺苷酸化位点(polyadenylation sites, PAS)及可变多聚腺苷酸化(alternative polyadenylation, APA)事件,对于深入揭示其生物学功能及生理病理的调控机制具有重要意义。传统的PAS鉴定方法一般依赖于特殊的3'序列富集测序(如3'-READS等),研究组前期开发了一种基于深度学习模型的单细胞精度PAS鉴定分析流程SCAPTURE(Li et al., Genome Biol, 2021),可从3'-tag单细胞测序数据中实现PAS位点的准确识别、筛选和定量。在这一项最新的研究中,团队进一步构建了物种特异的PAS位点分析流程SCAPTURE v2,利用海量3'-tag单细胞测序数据实现了多物种中单细胞精度的PAS位点准确识别,进一步揭示了物种间PAS位点的选择偏好,并据此构建了多物种PAS大百科全书数据库PASSpedia,其涵盖七物种、多组织的单细胞PAS位点搜索、浏览、分析、比较和下载等功能(图1),为PAS鉴定及其调控的相关研究提供了跨物种数据支持。
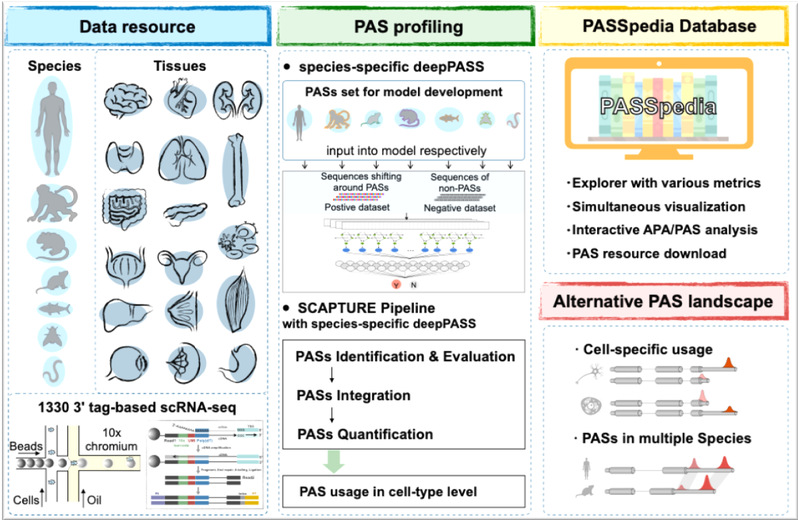
图1:PASSpedia数据库构建示意图
复旦大学生物医学研究院/复旦大学附属儿科医院杨力研究员和南芳博士后(现为中国科学院分子细胞科学卓越创新中心副研究员)为本文共同通讯作者。原中国科学院上海营养与健康研究所博士研究生张佩虹(现为美国芝加哥大学化学系博士后)、复旦大学附属儿科医院助理研究员冯华为本文共同第一作者。该项工作获得国家重点研发计划、国家自然科学基金、上海市科委的支持。
原文链接:https://doi.org/10.1093/gpbjnl/qzaf089