蓝斐团队报道H3K36me2 调控DNMT3A建立DNA甲基化的机制
DNA 甲基化作为一种重要的表观修饰,参与多种生物学过程,如异染色质形成、基因印记、X染色体失活、转座原件沉默等,近年来的研究也表明DNA甲基化异常与癌症发生密切相关,DNA甲基化紊乱成为癌症的一个新特征。
哺乳动物中,DNA甲基化主要发生在CpG上(5mC),这是由DNMT酶的特征决定的。人类基因组中约有80% CpG被甲基化,胚胎发育中DNA甲基化也高度动态变化,关于DNA甲基化建立和维持的调控机制目前仍是一个重要研究问题。已有研究证明组蛋白修饰可以调控DNA甲基化,如组蛋白H3K4零甲基化通过被ADD结构域识别诱导DNA甲基转移酶DNMT3A的活化构像以刺激其活性;而H3K4甲基化则不具备这种功能(表现为抑制信号),因此DNMT3A不作用于活跃启动子和增强子区域【1-3】;H3K36me3通过与DNMT3B的PWWP结构域结合招募DNMT3B促进活跃转录基因体区(gene body)的甲基化【4,5】,但基因间区的DNA甲基化调控过程尚不清楚。
近日,我院蓝斐研究员、吴飞珍副研究员和沈宏杰青年研究员合作在Protein & Cell 在线发表题为“DNMT3A Reads and Connects H3K36me2 to DNA Methylation”的短文,报道了DNMT3A可以通过识别H3K36me2 催化基因间区DNA甲基化建立这一机制,并在多发性骨髓瘤细胞系统中证明这一机制对于肿瘤增殖的潜在生物意义。

H3K36me2是哺乳动物基因组上含量很高的一种修饰,组蛋白质谱分析发现其丰度可达20%~40%,远远高于H3K36me3(约4-5%),广泛的分布在基因组中,包括基因间区以及部分活化基因区,但其生物学功能还不清楚。研究人员首先通过全基因组分析发现H3K36me2 和5mC在基因组上的分布高度相关(R=0.81),且二者在基因间区和启动子上游区域存在显著共定位,显著高于5mC与H3K36me3的相关性(R=0.63,主要集中在转录基因体区),提示H3K36me2 可能与DNA甲基化存在联系。体外生化实验发现DNMT3A的PWWP结构域对于H3K36me2有很强的识别(图2左),重要的是这一结合可以显著促进DNMT3A酶活(图2右)。这也是为数不多的组蛋白修饰直接激活DNA甲基转移酶酶活的机制(前述H3K4甲基化是抑制信号)。
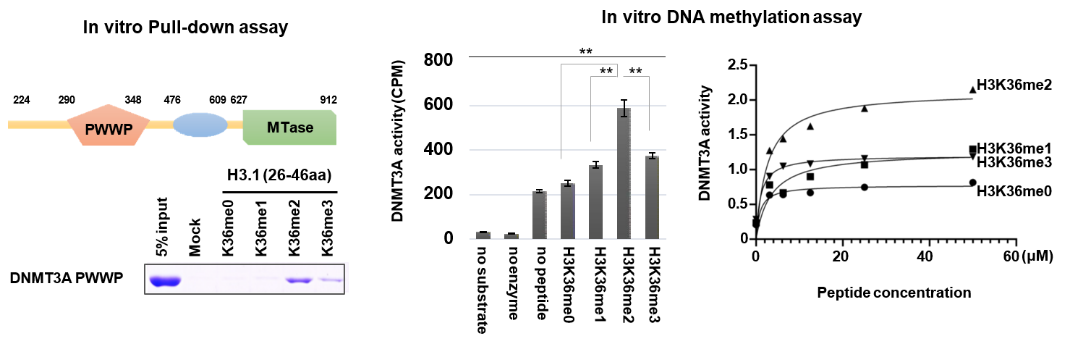
图1. DNMT3A通过PWWP domain识别H3K36me2且酶活增强
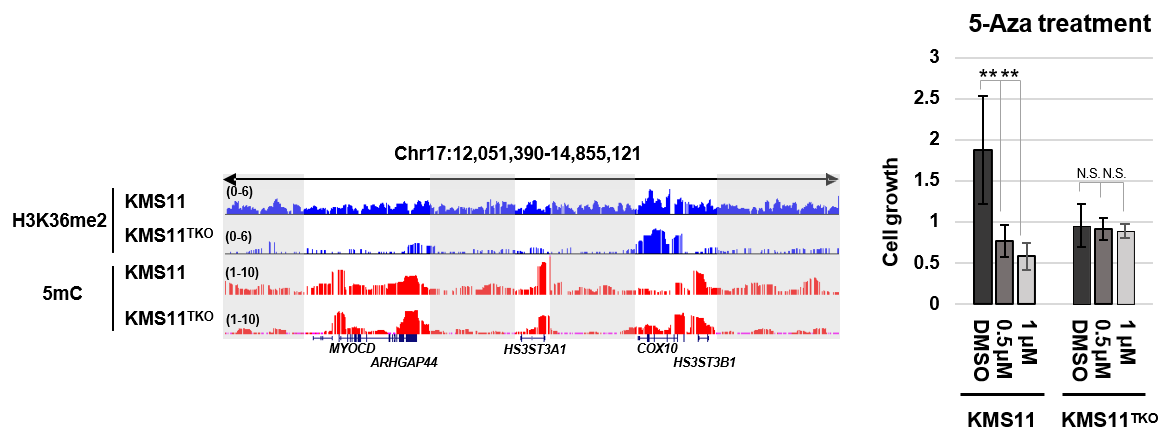
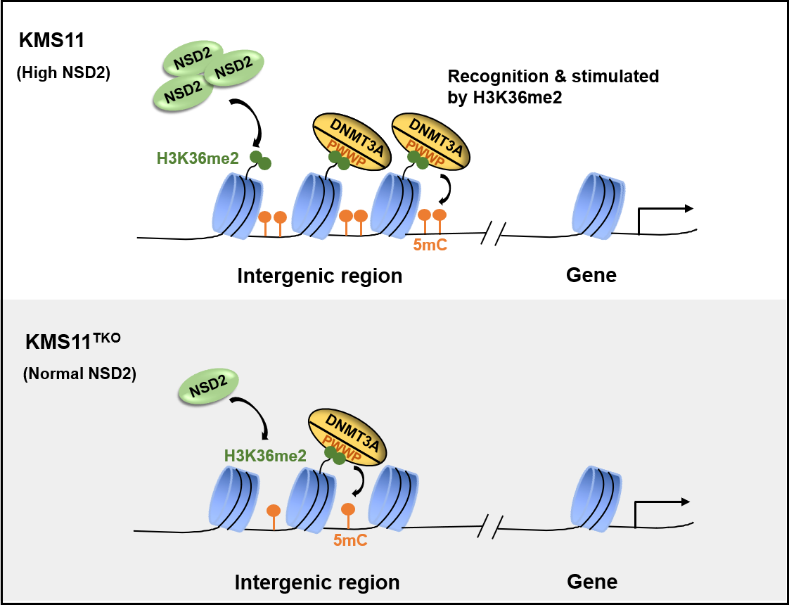
研究者进一步寻找此类调控的生理和病理意义。NSD家族甲基转移酶是H3K36me2主要的催化酶,它们常常在多类疾病中存在变异。例如,NSD2染色质易位过表达而引发的H3K36me2 水平升高是t(4;14)多发性骨髓瘤(multiple myeloma, MM)的主要致病原因。为了进一步研究H3K36me2对于5mC的可能调控机制及潜在生物意义,研究人员选取常用的KMS11 (含NSD2易位,H3K36me2异常高)和KMS11TKO(NSD2易位KO,仅有一个NSD2正常表达的拷贝,H3K36me2回复接近正常)细胞模型进行研究。发现KMS11中NSD2的过表达和高水平的H3K36me2导致了全基因组5mC水平的显著升高, 进一步通过ChIP-seq和MeDIP-seq分析对比KMS11和KMS11TKO的表观谱,发现这些异常的DNA高甲基化主要分布在基因间区(图2左)。最后,研究人员发现DNA甲基化抑制剂5-aza 处理可以特异性抑制KMS11细胞增殖而对KMS11TKO细胞影响较小,提示异常DNA甲基化抑制剂在t(4;14)多发性骨髓瘤治疗中的潜在价值(图2右)。
![]()
图2. KMS11细胞基因间区DNA甲基化水平升高,5-aza处理抑制其细胞增殖
需要提到的是,在本文投稿期间,哥伦比亚大学Chao Lu课题组2019年9月发表在Nature的文章“The histone mark H3K36me2 recruits DNMT3A and shapes the intergenic DNA methylation landscape”也报道了类似的发现【6】,H3K36me2通过招募DNMT3A可以调控基因间区DNA甲基化的建立。作者利用NSD1剂量不足导致的Sotos综合征病人血样和NSD1失活突变的头颈癌病人样本分析发现H3K36me2水平的降低会导致基因间区CpGme的丢失;同时,由DNMT3A胚系突变导致的TBRS综合征(一种儿童过度生长紊乱症)与Sotos综合征表现多种相同的临床症状,说明二者发病机制上的联系。体外生化实验发现三种点突变 (W297del,I310N和Y356C)导致DNMT3A的PWWP 结构域不能再识别并结合H3K36me2/3,测序分析进一步发现DNMT3A在基因组上的定位以及CpG甲基化·水平降低(主要是H3K36me2富集的基因间区),说明DNMT3A是通过PWWP结构域被H3K36me2招募至基因间区催化DNA甲基化建立的。此外,这篇文章中还提到H3K36me2和H3K36me3对于DNMT3A的竞争性结合,NSD1/2敲除后DNMT3A同样可以重新结合在H3K36me3富集的区域,可见H3K36甲基化修饰对于DNMT3A/B的染色质定位以及DNA甲基化调控的灵活性和多样性。
DNA甲基化与组蛋白修饰间的相互联系一直以来是表观遗传领域的一个主要研究问题,上述研究率先发现了H3K36me2对于基因间区DNA甲基化的调控,但基因组如此复杂,基因间区不同区域、不同调控元件又是具体如何受到调控的,以及这些不同区域的甲基化会如何影响基因表达,未来可能需要更多研究。
原文链接:https://doi.org/10.1007/s13238-019-00672-y
参考文献
【1】Reinhardt, R., and Fischle, W., et al.(2010). Chromatin methylation activity of Dnmt3a and Dnmt3a/3L is guided by interaction of the ADD domain with the histone H3 tail. NUCLEIC ACIDS RES 38, 4246-4253.
【2】Li, B.Z., Huang, Z., Cui, Q.Y., Song, X.H., Du L, Jeltsch, A., Chen, P., Li, G., Li, E., and Xu, G.L.(2011). Histone tails regulate DNA methylation by allosterically activating de novo methyltransferase. CELL RES 21, 1172-1181.
【3】Guo, X., Wang, L., Li, J., Ding, Z., Xiao, J., Yin, X., He, S., Shi, P., Dong, L., and Li, G., et al.(2015). Structural insight into autoinhibition and histone H3-induced activation of DNMT3A. NATURE 517, 640-644.
【4】Baubec, T., Colombo, D.F., Wirbelauer, C., Schmidt, J., Burger, L., Krebs, A.R., Akalin, A., and Schubeler, D.(2015). Genomic profiling of DNA methyltransferases reveals a role for DNMT3B in genic methylation. NATURE 520, 243-247.
【5】Neri, F., Rapelli, S., Krepelova, A., Incarnato, D., Parlato, C., Basile, G., Maldotti, M., Anselmi, F., and Oliviero, S.(2017). Intragenic DNA methylation prevents spurious transcription initiation. NATURE 543, 72-77.
【6】Weinberg, D. N., Papillon-Cavanagh, S., Chen, H., Yue, Y., Chen, X., Rajagopalan, K. N., . . . Lu, C. (2019). The histone mark H3K36me2 recruits DNMT3A and shapes the intergenic DNA methylation landscape. Nature, 573(7773).