蓝斐团队与合作者在《Cell Research》揭示减数分裂同源重组命运决定的表观遗传学基础
减数分裂为生殖细胞所特有的生物学事件,是生物有性生殖的基础。在减数分裂过程中,同源染色体的非姐妹染色单体间发生配对、联会和重组交换,而非同源染色体分配时自由组合,从而使配子呈现遗传多样化,增加了后代的适应性【1】。因此,减数分裂是保证物种繁衍、染色体数目稳定和物种适应环境变化而不断进化的基本前提。遗传变异是否与表观遗传调控有关是学术界长期关注的问题,本研究为回答该问题提供了一些重要线索。
同源重组是减数分裂的核心事件,它不仅是减数分裂过程中遗传物质交换的基础,也是同源染色体正确分离的保障。其过程高度复杂且受到严密调控。以小鼠为例,减数分裂同源重组起始于SPO11和GM960复合物所介导的DNA双链断裂 (Double-Stranded Breaks, DSBs);随后,断裂位点经5’末端切割、单链入侵等而使DSB获得修复;最终在同源染色体之间形成交叉(Crossover)或非交叉(Non-Crossover)【2】,其中,大部分DSBs修复形成非交叉,大约仅有10% DSBs修复生成交叉。
DSB位点并非随机分布,而是具有明显的偏好性,称之为热点(hotspot)。尽管已知PRDM9及其介导的H3K4me3修饰在在多数哺乳动物DSB热点选择中发挥着关键作用【3-5】,但是,关于PRDM9介导的H3K4me3在DSB修复过程中是如何演化的,PRDM9及其介导的H3K4me3是否参与调控DSB修复过程即同源重组命运决定,以及其它表观修饰因子是否也在其中发挥作用,等等,目前均不清楚。因为难以获取高纯度的各种亚型的哺乳动物减数分裂前期I细胞,所以同源重组命运决定机制一直是哺乳动物减数分裂研究的难点和热点。
2020年2月11日,我院蓝斐课题组与中科院分子细胞卓越创新中心(上海生物化学与细胞生物学研究所)童明汉课题组、北京大学汤富酬课题组合作在Cell Research上发表论文“Refined spatial temporal epigenomic profiling reveals intrinsic connection between PRDM9-mediated H3K4me3 and the fate of double-stranded breaks”。与之前PRDM9决定DSB热点研究不同,该文提出PRDM9及其介导的H3K4me3协同局部染色质环境可能参与了DSB修复过程,与同源重组命运决定相关;发现早期生成的DSBs更倾向于修复形成交叉。本文不仅为同源重组命运决定和交叉稳态的调控研究提供了新视角,也证实了遗传物质交换机制和表观遗传调控的相关性。

得益于童明汉课题组前期建立的分离小鼠任意类型生精细胞的技术体系,使获取高纯度同步发育的任意类型生精细胞成为现实,因此有可能对精子发生过程中关键生物学事件的分子机制进行精细研究。比如,童明汉课题组曾经基于上述体系与汤富酬、李劲松课题组合作,2018年7月30日在Cell Research上发表了题为“Single-cell RNA-seq uncovers dynamic processes and critical regulators in mouse spermatogenesis”的论文,绘制了精子发生过程中生精细胞转录组的动态变化图谱,系统地阐明了这种动态变化的可能分子基础,为哺乳动物精子发生调控的分子机制研究提供了基础数据【6】。
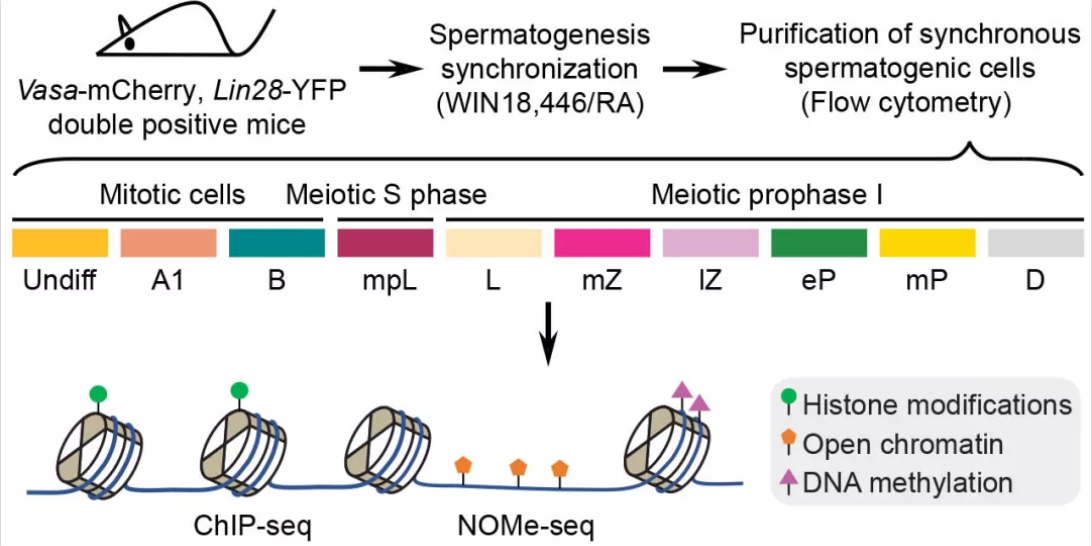
图1: 基于精细分期的减数分裂重组过程表观遗传组的建立
作为减数分裂系列研究的一部分,为了阐明表观遗传是否以及如何调控减数分裂同源重组,本文在纯化各级生精细胞的基础上,将减数分裂前期I细胞细分为7个时期进行研究(图1),结合ChIP-seq技术,分析了包括H3K4me3在内的8种组蛋白修饰在减数分裂前后的动态变化;应用NOMe-seq,观察了DNA甲基化以及染色质开放状态的动态变化(图1),揭示了之前难以发现的减数分裂前期I细胞演化的未知规律。
结果显示,PRDM9介导的H3K4me3修饰在细线期(Leptotene)开始生成,至偶线期中期达到峰值,随后逐步消失(图2a);同时,越早产生的H3K4me3越晚消失。这种H3K4me3动态变化与DSB修复过程中同源重组中间产物的演变高度相关,提示PRDM9介导的H3K4me3可能参与了DSB修复过程。根据生成和消失时间,研究人员继而将与重组热点相关的H3K4me3分为四种类型,发现早期产生的H3K4me3信号强度与宽度均最强,且富集了更长的PRDM9结合基序(图2b-e);那些快速消失的H3K4me3信号强度与宽度均最弱,且为较晚产生。这些结果表明早期生成的PRDM9介导的H3K4me3可能通过影响同源重组稳定中间体dHJ(double Holliday Junction),从而进一步调控交叉形成;而那些弱而快速消失的则指导生成非交叉。
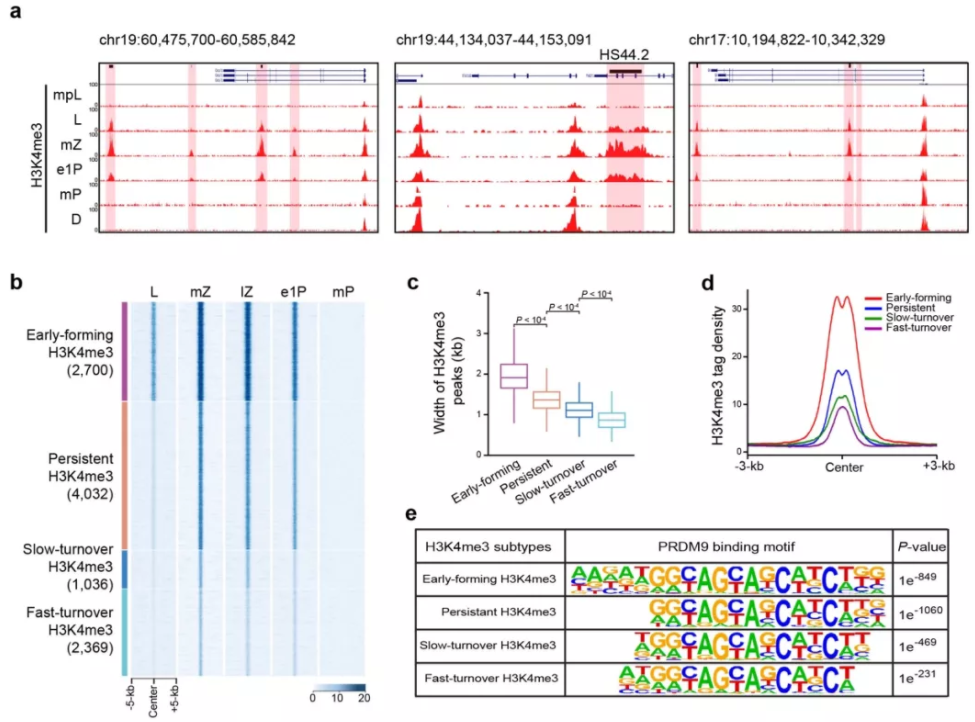
图2: PRDM9介导的H3K4me3修饰的特性a. PRDM9介导的H3K4me3代表性UCSC track,浅红色背景指示PRDM9介导的H3K4me3信号;b. 热图展示了H3K4me3信号强度,依据H3K4me3修饰的周转时间将其分为四种类型;c-d. mZ阶段H3K4me3修饰信号的平均宽度与强度在上述四种类型中的比较;e. PRDM9结合基序在上述四种类型中的富集情况。
与H3K4me3相比,H3K4me1修饰在重组热点上出现得更早,消失得更晚,并且覆盖了更多的核小体。有意思的是,在偶线期阶段可以明显观察到DSB核心区域核小体H3K4me1被H3K4me3所替代的情况。鉴于体外实验证实PRDM9具有催化H3K4me1的活性,研究人员推测H3K4me1可能是PRDM9催化产生H3K4me3过程的中间态;而早出现的H3K4me1则提示重组热点决定发生的时间可能更早。
除上述修饰外,本研究还发现了H3K36me3与H3K27ac也富集于重组热点,而H3K9me2修饰则缺失于重组热点;同时,观察到重组热点区域的染色质与全基因组相比更加开放。有意思的是,不论H3K36me3与H3K27ac的富集,还是H3K9me2的缺失,抑或染色质的开放,均随Prdm9敲除而变化,表明上述表观因子均为PRDM9依赖性的。与H3K4me3类似,相比于其它类型,早期产生的H3K36me3与H3K27ac具有更强的信号强度与宽度,染色质也最为开放,提示H3K36me3, H3K27ac以及染色质开放可能与H3K4me3一起共同在DSB命运决定中发挥调控作用。同时,研究者根据所上述表观遗传规律,发现了4537个潜在的DSB热点,其中置信度最高的302个位点值得后续进行深入的机理分析。
综上所述,本研究系统地阐述了PRDM9介导的H3K4me3以及H3K4me1、H3K36me3、H3K27ac、H3K9me2和染色质开放在减数分裂过程及其前后的动态变化,阐明了PRDM9及其介导的H3K4me3修饰与DSB命运决定的相关性,提出了“PRDM9及其介导的H3K4me3修饰不但能决定DSB位点选择,而且可能调控DSB形成后的修复过程,从而决定DSB命运”的假说。该假说将是相关团队的下一个重点研究目标。
据悉,中国科学院分子细胞卓越创新中心(上海生物化学与细胞生物学研究所)陈瑶博士,复旦大学生物医学研究院吕瑞途博士、荣博文博士生,北京大学未来基因诊断高精尖创新中心博士生郑宇轩与中国科学院分子细胞卓越创新中心(上海生物化学与细胞生物学研究所)林震博士为共同第一作者。蓝斐研究员、中科院分子细胞卓越创新中心(上海生物化学与细胞生物学研究所)童明汉研究员与北京大学汤富酬研究员为文章共同通讯作者。
原文链接:
https://www.nature.com/articles/s41422-020-0281-1.pdf
参考文献
1. Baudat, F., Imai, Y. & de Massy, B. Meiotic recombination in mammals: localization and regulation. Nat Rev Genet 14, 794-806, doi:10.1038/nrg3573 (2013).
2. Hunter, N. Meiotic Recombination: The Essence of Heredity. Cold Spring Harb Perspect Biol 7, doi:10.1101/cshperspect.a016618 (2015).
3. Baudat, F. et al. PRDM9 is a major determinant of meiotic recombination hotspots in humans and mice. Science 327, 836-840, doi:10.1126/science.1183439 (2010).
4. Myers, S. et al. Drive against hotspot motifs in primates implicates the PRDM9 gene in meiotic recombination. Science 327, 876-879, doi:10.1126/science.1182363 (2010).
5. Parvanov, E. D., Petkov, P. M. & Paigen, K. Prdm9 controls activation of mammalian recombination hotspots. Science 327, 835, doi:10.1126/science.1181495 (2010).
6. Chen, Y. et al. Single-cell RNA-seq uncovers dynamic processes and critical regulators in mouse spermatogenesis. Cell Res 28, 879-896, doi:10.1038/s41422-018-0074-y (2018).