曹纬倩与合作者《Nature Methods》发布蛋白质位点特异性糖基化解析工具pGlyco3
蛋白质位点特异性糖基化的精准解析对于研究蛋白质糖基化功能、发现疾病潜在标志物及药物靶标十分重要。大规模、高准确性完整糖肽解析一直是蛋白质后修饰分析领域极具挑战性的工作。近年来,完整糖肽解析在生物质谱分析及计算糖蛋白质组领域都取得了重要进展;然而,目前解析方法仍有明显的局限性:大部分工具无法对糖链鉴定进行质控,尤其是复杂的糖型;检索速度慢;无法对多个糖基化位点精准定位;无法鉴定含有修饰糖单元的完整糖肽等问题。
2012年11月25日,我院曹纬倩博士与中科院计算所曾文锋博士等合作在Nature Methods上在线发表了题为“Precise, Fast and Comprehensive Analysis of Intact Glycopeptides and Modified Saccharide Units with pGlyco3”的研究论文。文章介绍了一种可用于完整糖肽及修饰糖精准、快速解析的工具pGlyco3。该工具提出了糖链优先的搜索策略(glycan-first search),大大提高了鉴定精度和检索速度。

当前大部分解析工具都是通过修改常规蛋白质搜索引擎,使得引擎能够支持糖链的检索。但是这些软件不容易对糖链鉴定进行质控,尤其是复杂的糖型。研究者提出了糖链优先的检索策略,其特色就是先搜索糖链部分,通过糖链鉴定的质控,缩小搜索空间从而提高检索速度,并且过滤掉不可靠的糖型从而提高鉴定精度。
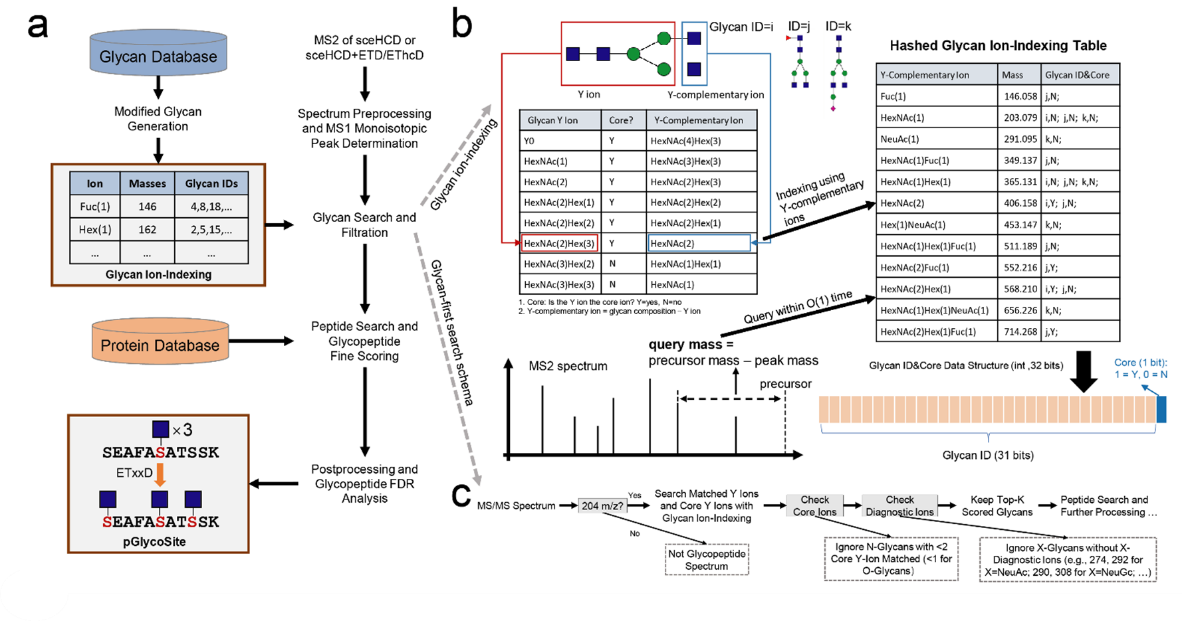
图1.pGlyco3解析流程。图片来源 Nat Methods
研究人员采用他们前期开发的基于重标技术的糖肽鉴定及陷阱库检索的质控流程,在复杂样本酵母中,客观地比较了不同检索引擎汇报糖肽结果的假阳性率(包括常见糖肽检索引擎Byonic,O-Pair和MSFragger-Glyco),同时比较了不同检索引擎的检索速度,证明pGlyco3不管是在鉴定准确性还是检索速度方面都有更好的表现。
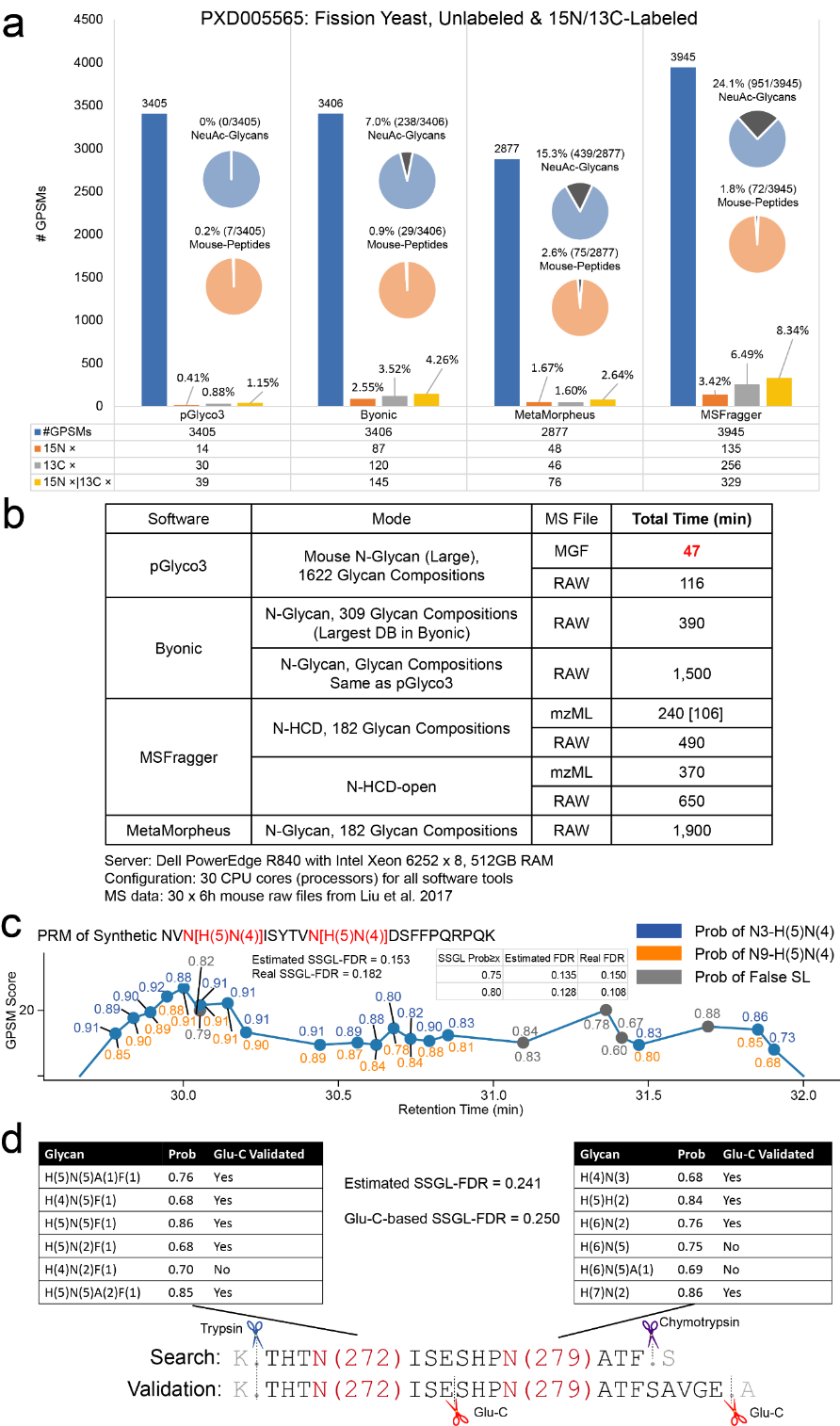
图2.pGlyco3鉴定结果比较和位点准确性验证。图片来源 Nat Methods
此外,pGlyco3糖库中糖链采用线性编码,用户可以根据经验构建样品特异性的糖库,进行完整糖肽上含有修饰糖单元的鉴定。采用该方法,研究者在他们的酵母数据中发现了大量(约50%)的氨基修饰的糖单元,以及部分磷酸修饰的糖单元;在其它公开的数据中也发现了氨基修饰的糖单元。
对于含有多个糖基化修饰位点的糖肽,pGlyco3采用动态规划算法得到最优匹配路径,并且估计最优匹配为随机匹配的概率,从而实现糖基化位点的精准定位。与已有的位点特异性糖链定位算法相比,pGlyco3的定位速度和精度都有大幅提升。同时研究人员分别采用合成的含双糖基化位点的标准完整糖肽,以及多种酶切策略对位点鉴定的准确性进行了有效验证。
pGlyco3是继该团队发展的pGlyco 2.0(Nat Commun,2017)后的又一重要突破,解决了基于数据库检索的完整糖肽解析中面临的准确性、多位点定位、修饰糖鉴定的重要难题,为糖蛋白质组分析领域提供了重要的鉴定策略和检索工具,有望促进蛋白质糖基化相关功能和生物标志物的研究。
据悉,北京中科院计算所毕业的曾文锋博士(现为德国马克斯普朗克生物化学研究所博士后)和我院曹纬倩副研究员为该论文共同第一作者。曾文锋博士为通讯作者,同时计算所贺思敏研究员和我院杨芃原教授(1949.6.12-2021.5.31)共同指导了本研究项目的开展;刘铭琪博士做出了重要贡献。该项目得到国家重点研发计划,国家自然科学基金委重大研究计划培育项目,以及上海市高水平地方高校创新研究团队的支持。
原文链接:https://www.nature.com/articles/s41592-021-01306-0