Cell Research|陈飞团队揭示出H3K4me2/3直接调控基因转录的机制
基因表达的精准调控,对机体发育和细胞的各种生理功能的维持至关重要。真核生物基因的转录主要分为转录起始(initiation)、暂停-释放(pausing-release)、延伸(elongation)和终止(termination)四个步骤,转录进程的顺利进行与DNA修饰、组蛋白修饰和RNA修饰等表观遗传修饰高度相关【1】。组蛋白修饰(histone modification)是指组蛋白在相关酶催化下发生甲基化、乙酰化、磷酸化、泛素化和ADP核糖基化等动态修饰的过程,它可通过招募效应蛋白来改变染色质的开放或凝聚的状态,进而调控基因的表达。作为表观遗传学中最为重要和多样的一种调控方式,它的失调与多种疾病如肿瘤的发生发展密切相关。因此,理解组蛋白修饰并阐明其发挥作用的机理对领域的发展至关重要。
组蛋白H3第4位赖氨酸的甲基化修饰(H3K4)在进化上高度保守,是被研究最多的组蛋白修饰。在酵母中,SET1蛋白是发现最早且唯一的H3K4甲基转移酶,含有SET1的复合物被命名为COMPASS(COMplex of Proteins Associated with Set1)。在哺乳动物中,SET1有六个同源蛋白—SET1A、SET1B、MLL1、MLL2、MLL3和MLL4,H3K4的甲基化修饰由这6个COMPASS复合物介导。H3K4me3在转录起始位点的富集程度与基因表达水平息息相关,体外生化实验证明H3K4me3能与转录起始复合物TFIID中的TAF3 PHD结构域结合,进而促进基因转录的起始【2,3】。因此,H3K4me3一直被认为是通过促进转录的起始来调控基因的转录水平。
随着研究的推进,领域内对COMPASS催化活性的功能也产生了一些疑问。如小鼠胚胎干细胞的增殖和自我更新不需要SETD1A的催化活性【4】,MLL3/4蛋白对小鼠胚胎的存活及发育是必须的,但它们催化的H3K4me1修饰对增强子的激活却是可有可无的5。在酿酒酵母中,敲除甲基转移酶复合物COMPASS的组分Spp1后,H3K4me3的含量显著下降,但新生RNA或总RNA都没显著差异【6,7】。不禁让人好奇:H3K4修饰对转录是否具有因果关系,具体的机制又是什么?
2023年3月15日,复旦大学生物医学研究院陈飞团队在Cell Research上发表了题为H3K4me2/3 modulate the stability of RNA polymerase II pausing的研究论文,回答了困扰领域20多年的科学问题。发现H3K4me2/3在细胞内是通过维持暂停Pol II(paused Pol II)在近端启动子区的稳定而促进基因激活;而H3K4me2/3对转录起始没有明显的调控作用。

大量的生化和结构生物学的研究表明:WDR5、ASH2L、RBBP5、DPY30等组分是哺乳动物中6个COMPASS复合物的共有组分,它们对COMPASS和甲基转移酶活性维持至关重要。dTAG蛋白快速降解系统是研究转录调控机制的有力工具。为了研究H3K4甲基化修饰对转录是否具有直接的调控作用,研究人员在小鼠胚胎干细胞(mESCs)中利用CRISPR-Cas9系统构建了FKBP12F36V-RBBP5和DPY30(RBBP5-dTAG和DPY30-dTAG)细胞系,从而可以对内源蛋白进行快速降解。令人意外的是,RBBP5和DPY30降解6小时后H3K4me2/3的甲基化程度显著减少,这与H3K4甲基化比较稳定的传统观点不一致;这一过程可被pan-KDM5抑制剂逆转,表明H3K4me2/3甲基化的减少是由KDM5的去甲基化作用介导的。随着蛋白降解时间的延长,约12-24小时可清除H3K4me3和H3K4me2,需要更长的时间才可能清除H3K4me1,表明H3K4me2/3相对于H3K4me1更加动态(图1)。ChIP-Rx结果显示蛋白降解6小时后H3K4me3在全基因组上的分布均显著降低,大部分H3K4me2在全基因组上的分布也显著降低,变化趋势与WB显示的结果一致。
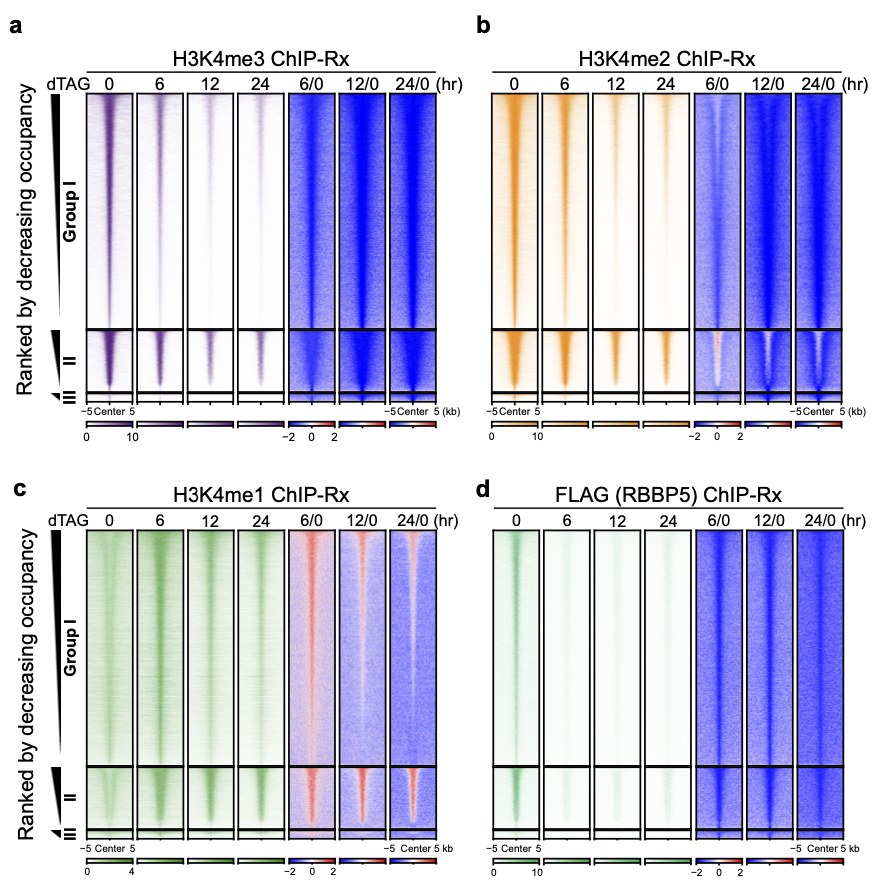
图1 RBBP5蛋白快速降解后组蛋白修饰变化谱
为了探究H3K4me2/3的减少是否会影响转录,研究人员在蛋白降解不同时间的mRBBP5-dTAG小鼠胚胎干细胞系中进行Pol II ChIP-Rx,发现RBBP5降解6小时后全基因组启动子区Pol II的分布开始略微减少,当蛋白降解12小时后发现Pol II在启动子区的分布降低极为显著。有意思的是,Pol II在启动子区上下降的速率与H3K4me2/3下降的速率有个明显的时间差。当RBBP5降解6小时后H3K4me2/3在启动子区上的分布已经显著下降,而Pol II的分布则开始略微减少;当RBBP5降解12小时后H3K4me2/3接近被清除干净时,Pol II在启动子区上的分布则下降显著(图2a),并且这种降低能被pan-KDM5抑制剂回补。这些结果说明H3K4me2/3具有维持启动子区Pol II稳定的作用。
启动子区Pol II由转录起始复合物(PIC)Pol II和暂停Pol II(paused Pol II)构成,PIC Pol II可通过转位酶XPB/TFIIH的作用变成暂停Pol II,这一过程可被雷公藤甲素(Triptolide)抑制。为了回答H3K4me2/3具体维持哪类Pol II的稳定,研究人员用Triptolide处理RBBP5蛋白降解12小时后的细胞,发现PIC Pol II在启动子区的分布没有减少,揭示了H3K4me2/3具有维持启动子区暂停Pol II稳定的作用。PRO-seq (Precision nuclear run-on sequencing)技术能在单个碱基分辨率检测具有转录活性Pol II的定位,利用该技术分析RBBP5蛋白快速降解后Pol II的分布,发现基因启动子暂停Pol II也显著减少(图2b),基本没观察到转录暂停-释放的现象。
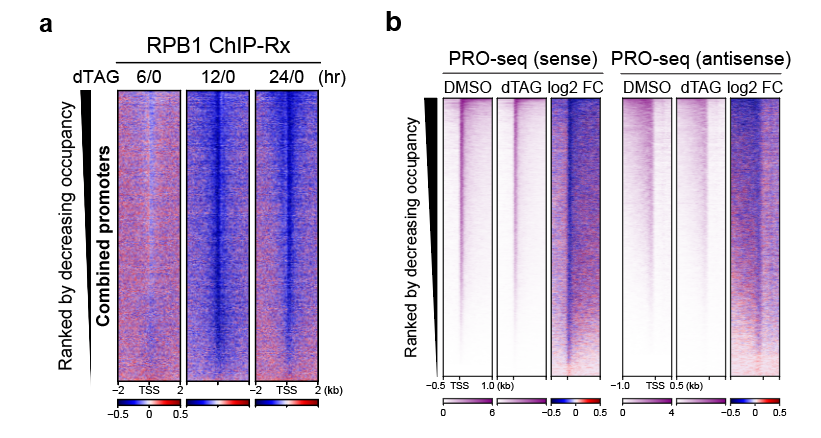
图2 H3K4me2/3对基因转录调控的作用
暂停Pol II可能通过暂停-释放而继续转录延伸并合成RNA(productive elongation),也可能通过启动子近端终止(promoter-proximal termination)的方式从染色质上的启动子附近解离。为了明确暂停Pol II的命运,研究人员利用ChIP-Rx分析了Pol II 在转录起始位点(TSS)和转录终止位点(TES)间的分布,发现RBBP5蛋白快速降解后Pol II 在gene bodies内的分布也是下降的。分别分析Pol II ChIP-Rx和PRO-seq两套数据,均发现启动子区域Pol II下降的程度与gene bodies内Pol II的减少呈正相关,即启动子区Pol II变化的趋势与gene bodies内Pol II的变化一致。这些结果表明启动子暂停Pol II的减少不是由于转录暂停-释放增加造成的。
长久以来,人们认为H3K4me3通过招募TFIID来促进转录起始复合物(PIC)的组装,进而促进基因转录。为了回答启动子区Pol II的减少是否由转录起始缺陷导致的,利用ChIP-Rx在RBBP5-dTAG细胞系中检测TAF1(TFIID复合物的支架蛋白)和TAF3(直接识别H3K4me3)在全基因组上的分布。发现当RBBP5蛋白降解24小时后TAF1和TAF3在转录起始位点(TSS)上的分布没有减少,而此时H3K4me2/3基本被清除干净。H3K4me2/3减少导致启动子区Pol II的减少与转录起始无关。
综上所述,该工作利用小鼠胚胎干细胞模型研究了H3K4me2/3对基因转录的直接调控作用,回答了长期困扰领域的问题。发现H3K4me2/3不影响转录的起始或调控转录的暂停-释放,而是通过维持暂停Pol II在近端启动子区稳定的新颖机制来调控转录。为研究H3K4me2/3在细胞命运决定和病变进展的作用机制提供全新视角。
复旦大学附属肿瘤医院助理研究员胡士斌、生物医学研究院博士生宋爱霞和肿瘤医院博士后彭林娜为本文共同第一作者,唐楠、乔志斌和王振宁博士对本文也有重要的贡献,生物医学研究院/附属肿瘤医院的陈飞和胡士斌为本文的通讯作者。本课题还得到蓝斐实验室的荣博文博士和何晨曦博士的帮助。
值得一提的是,近日美国纪念斯隆-凯瑟琳癌症中心Kristian Helin研究组在Nature上发表了题为H3K4me3 regulates RNA polymerase II promoter-proximal pause-release的研究论文【8】,对H3K4me3在基因转录暂停和释放中的调控功能进行了报道。该论文同样发现了H3K4me3对转录暂停而非转录起始的直接调控作用。
原文链接:
https://www.nature.com/articles/s41422-023-00794-3
参考文献
1 Chen, F. X., Smith, E. R. & Shilatifard, A. Born to run: control of transcription elongation by RNA polymerase II. Nat Rev Mol Cell Biol 19, 464-478, doi:10.1038/s41580-018-0010-5 (2018).
2 Lauberth, S. M. et al. H3K4me3 interactions with TAF3 regulate preinitiation complex assembly and selective gene activation. Cell 152, 1021-1036, doi:10.1016/j.cell.2013.01.052 (2013).
3 Vermeulen, M. et al. Selective anchoring of TFIID to nucleosomes by trimethylation of histone H3 lysine 4. Cell 131, 58-69, doi:10.1016/j.cell.2007.08.016 (2007).
4 Sze, C. C. et al. Histone H3K4 methylation-dependent and -independent functions of Set1A/COMPASS in embryonic stem cell self-renewal and differentiation. Genes Dev 31, 1732-1737, doi:10.1101/gad.303768.117 (2017).
5 Dorighi, K. M. et al. Mll3 and Mll4 Facilitate Enhancer RNA Synthesis and Transcription from Promoters Independently of H3K4 Monomethylation. Mol Cell 66, 568-576 e564, doi:10.1016/j.molcel.2017.04.018 (2017).
6 Murray, S. C. et al. H3K4me3 is neither instructive for, nor informed by, transcription. bioRxiv, 709014, doi:10.1101/709014 (2019).
7 Howe, F. S., Fischl, H., Murray, S. C. & Mellor, J. Is H3K4me3 instructive for transcription activation? Bioessays39, 1-12, doi:10.1002/bies.201600095 (2017).
8 Wang, H. et al. H3K4me3 regulates RNA polymerase II promoter-proximal pause-release. Nature 615, 339-348, doi:10.1038/s41586-023-05780-8 (2023).