Nature Metabolism 丨王罡琦等开发空间定量代谢组学检测方法并揭示脑卒中后大脑代谢重塑
空间生物学已成为生命科学和生物技术研究的前沿领域。其中,空间代谢组学作为关键分支,能够揭示代谢物及其微环境的原位分布。质谱成像(MSI)是目前研究代谢物空间分布的主要技术手段,但其定量分析仍面临诸多挑战,如基质效应、加合物形成和源内碎裂等【1,2】。这些问题是质谱本身的固有限制,影响了两大主流MSI技术——基质辅助激光解吸电离(MALDI)和解吸电喷雾电离(DESI)【2,3】,不仅妨碍组织间比较,更严重的是限制了同一组织内不同区域间的差异分析。
例如,在脑组织中均匀喷涂脂质内标 PE15:0/18:1 (d7) 时,使用 norharman 和 2,5-二羟基苯甲酸(2,5-DHB)两种不同基质所得到的MALDI-MSI图像在峰值强度分布上差异显著【4】。在小鼠胶质母细胞瘤模型中,MALDI-MSI 与 DESI-MSI 对棕榈酸和硬脂酸的检测结果在肿瘤与正常区域间也呈现相反趋势【5】。此外,实验条件和仪器日常波动导致的批次效应,也进一步削弱了数据的可比性【6】。因此,空间代谢组学的定量化和标准化至今仍未得到根本解决,导致MSI结果存在“眼见未必为实”的问题。
目前,最常用的定量策略是使用同位素标记的内标(IS),该方法已成功应用于空间脂质组学研究,能够有效校正基质效应并提高实验可重复性【4】。然而,将其拓展至代谢组学仍面临困难。与结构相对一致的脂质不同,水溶性代谢物具有更复杂且多样的物理化学性质,即使同属一个代谢类别,其结构和行为也可能差异巨大,因此通常需要针对每种代谢物设计专属内标。然而,大规模构建这种内标面板不仅成本高昂,还常受限于可获得性。
2025年8月5日,复旦大学生物医学研究院/复旦大学附属儿科医院王罡琦团队在Nature Metabolism发表研究论文Spatial quantitative metabolomics enables identification of remote and sustained ipsilateral cortical metabolic reprogramming after stroke。该研究提出一种将¹³C标记酵母提取物作为内标,并结合MALDI-MSI的空间定量代谢组学方法,可实现200余种代谢特征的像素级归一化与相对定量,部分代谢物甚至可实现绝对定量。

该方法基于已验证的“组织表面喷涂”策略:将¹³C标记的酵母提取物均匀喷涂于热灭活组织表面,再喷涂N-(1-萘基)乙二胺二盐酸盐(NEDC)基质,随后使用TimsTOF flex MALDI2质谱仪在负离子模式下进行检测(图1)。喷涂内标前后,组织样本的质谱质量相当。经去除部分信号重叠峰后,在肾脏和脑组织中分别识别出171个和170个可用于像素级归一化的代谢分子,其中145个可应用于两种组织。同时,还鉴定出多种¹³C标记脂质类内标,用于校正相应类别的脂质分子。最终,在脑组织中共定量了8类脂质的146个脂质分子,以及131个代谢产物。
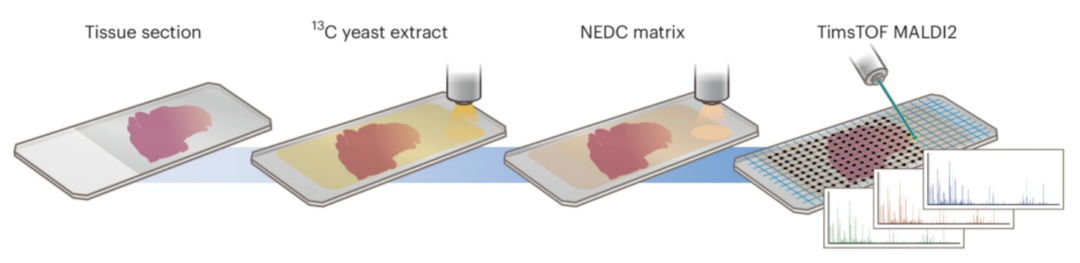
图1 空间定量代谢组学方法流程图
研究以小鼠脑卒中模型为例,描绘了梗死核心区及周边区域的代谢重编程过程。首次发现,组织学上未受损的同侧皮层也发生了显著的代谢变化。在卒中后第7天,该区域赖氨酸水平升高(具有神经保护作用),而谷氨酸水平降低。至第28天,赖氨酸和谷氨酸恢复至基线水平,但与易损性相关的代谢前体UDP-GlcNAc和亚油酸仍持续处于低水平,提示该区域存在长期、潜在有害的代谢重塑。有趣的是,若不使用内标,仅靠传统的归一化策略,无法识别这些关键差异。
该研究显著提升了空间代谢组学的定量准确性,为其在生物医学研究和临床诊断中的应用奠定了坚实基础。研究团队也指出,尽管本研究已实现部分代谢物的绝对定量,但代谢物注释的不确定性仍是当前的一大挑战。
复旦大学生物医学研究院、复旦大学附属儿科医院王罡琦研究员为本文第一作者和共同通讯作者,荷兰莱顿大学医学中心Ton Rabelink教授和Martin Geria教授为共同通讯作者。
原文链接:https://www.nature.com/articles/s42255-025-01340-8
参考文献:
1. Tobias, F. & Hummon, A. B. Considerations for MALDI-based quantitative mass spectrometry imaging studies. J. Proteome Res.19, 3620–3630 (2020).
2. Lanekoff, I., Stevens, S. L., Stenzel-Poore, M. P. & Laskin, J. Matrix effects in biological mass spectrometry imaging: identification andcompensation. Analyst139, 3528–3532 (2014).
3. Taylor, A. J., Dexter, A. & Bunch, J. Exploring ion suppression in mass spectrometry imaging of a heterogeneous tissue. Anal. Chem.90, 5637–5645 (2018).
4. Vandenbosch, M. et al. Toward omics-scale quantitative mass spectrometry imaging of lipids in brain tissue using a multiclass internal standard mixture. Anal. Chem.95, 18719–18730 (2023).
5. Schwaiger-Haber, M. et al. Using mass spectrometry imaging to map fluxes quantitatively in the tumor ecosystem. Nat. Commun.14, 2876 (2023).
6. Balluff, B., Hopf, C., Porta Siegel, T., Grabsch, H. I. & Heeren, R. M. A. Batch effects in MALDI mass spectrometry imaging. J. Am. Soc. Mass. Spectrom.32, 628–635 (2021).