顾宏周课题组《Chem》报道利用DNA核酶开发高效制备单链DNA纳米结构的新方法
近15年来,作为构建DNA纳米组装体的主要方法之一,DNA折纸术(DNA origami)可使得一条长单链DNA在成百或千条短链DNA的辅助下, 通过碱基互补配对原则折叠并锁定生成所设计的纳米图案,它的出现极大地推动了DNA纳米技术领域的发展。单链DNA折纸术(single-stranded DNA origami)是传统DNA折纸术的一种进化和衍生,它摒除了对众多短序列的需求,通过高度集成序列信息于一条长单链DNA中, 实现了单个DNA序列的自我折叠和组装体构建(类似于蛋白质由一条长肽链折叠成三维结构)。
单链DNA组装体(ssDNs)的可编程性和可寻址性均能与传统的多链DNA 组装体相媲美,此外它们还具有产物单一、纯净度高的先天优势及可在生物体内复制的潜力。然而,目前对于200个碱基以上的DNA序列,化学合成的成本高、产量低,当序列中包含较多的二级结构时会进一步阻碍合成的效率,这些因素限制了单链DNA组装体在生命和材料科学等领域的应用和发展。
2021年3月30日,复旦大学生物医学研究院顾宏周课题组在Chem杂志上发表题为“DNA-catalyzed efficient production of single-stranded DNA nanostructures”的工作,报道了一种高效制备单链DNA纳米结构的生物方法。作者通过将多个单链DNA组装体序列以假基因片段形式与自切割型DNA核酶序列有机串联成一个整体,并重组到噬菌体基因组中,实现了依托噬菌体存储组装体序列信息,可随时扩增放大并根据需求制备相应单链DNA组装体的策略。

作者以一条由520 个碱基的序列折叠而成的三角形组装体为例,展示了制备方法的整个流程(图1)。首先,通过电脑程序辅助设计出可自组装成三角形的长单链DNA序列;随后,把该组装体序列作为一个假基因片段进行化学合成,并在片段的两端各自插入一个Zn2+依赖的自切割型DNA核酶(有催化能力的核酸序列);接着,将该片段重组到质粒或噬菌粒中,通过体外或体内的滾环复制扩增方式放大制备单链DNA序列;最后,在添加Zn2+的条件下,DNA核酶自发切割并释放出单链DNA组装体序列。
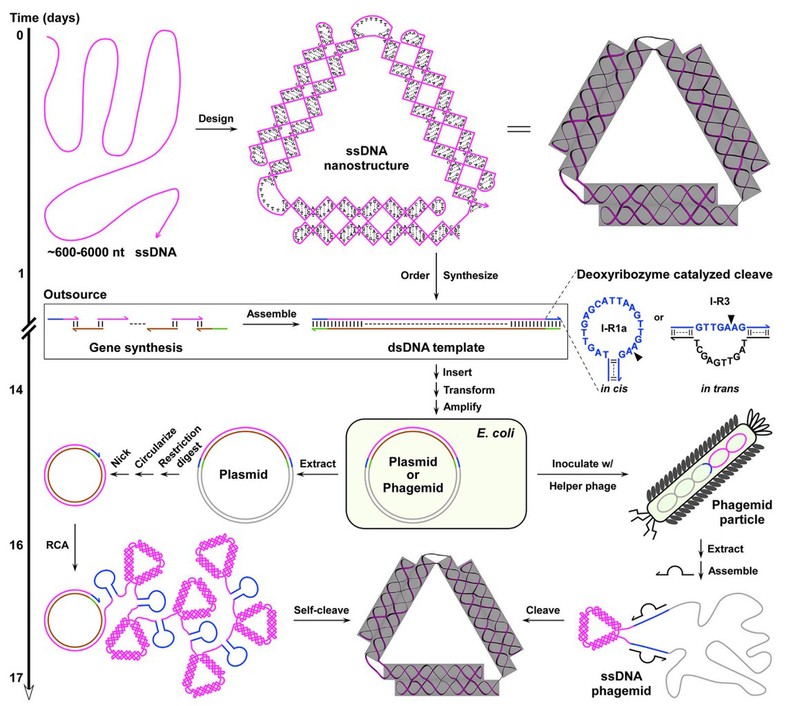
图1利用自切割型DNA核酶制备单链DNA组装体的示意图和时间表
在德国人Hendrik Dietz教授团队“Biotechnological mass production of DNA origami”(Nature,2017)文章中利用顺式(cis)DNA核酶制备单链DNA的基础上,作者拓展了活性更强的反式(trans)核酶的使用。当多种不同形状(三角形,520 nt;四边形,680 nt;菱形,2200 nt;方形结,1700 nt)的单链DNA组装体出现时有所需的状况时(图2),作者提出以串联多个组装体和反式核酶序列的方式,既能在重组和放大过程中节约时间和劳动力,又能在切割分离目的组装体时获得高效性和可控性。依托基于反式核酶的生物法,作者制备单链DNA组装体时成本可低至1500 rmb/g,产量可达g-kg,长度可至10000个碱基以上。
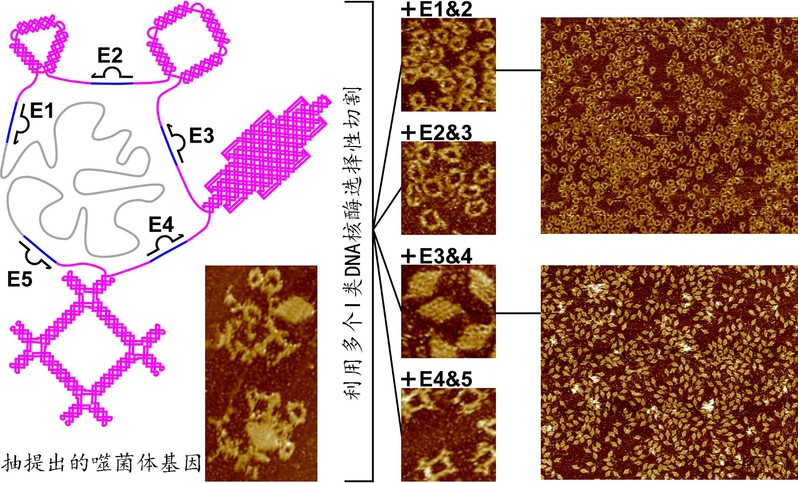
图2利用反式(trans)切割型DNA核酶高效可控地分离制备单链DNA组装体
综上,该研究提出了一种利用反式切割型DNA核酶突破化学合成长单链DNA序列瓶颈的生物法策略,为单链DNA组装体的高效制备奠定了基础,也为基于DNA组装体的下游应用,如辅助脂质体分离制备及研究细胞对脂质体的摄取等扫清了障碍。此外,长单链DNA序列的高效制备还可能为基于长程单链DNA片段的基因敲入、基于长程锁式探针的长程测序等新技术的发展提供支撑。
据悉,复旦大学生物医学研究院顾宏周研究员(复旦大学附属口腔医院双聘研究员)为本文通讯作者。贾友礼、陈黎曼为本文的共同第一作者。
原文链接:https://www.cell.com/chem/fulltext/S2451-9294(20)30633-1