Briefings in Bioinformatics丨杨力团队开发RNAlight工具用于多类型RNA的亚细胞定位预测研究
RNA亚细胞定位与其生成、加工和功能密切相关(1,2),因此,解析RNA的亚细胞定位对其功能研究至关重要。利用传统的实验生物学方法,如FISH和细胞组分分离后鉴定等可以有效的发现目的RNA的亚细胞定位,但是其通量较低、且不同实验方法的特异性也会导致某些RNA无法被准确定位。近期,结合亚细胞RNA组分分离和后续高通量测序分析,大量的RNA亚细胞定位信息被报道,利用这些数据并结合机器学习和深度学习的方法,也实现了对RNA亚细胞定位的预测,但是已有的预测方法仅局限于单一类型的RNA分子,并且模型可解释性较差,未能提供影响RNA亚细胞定位的关键信息。
2022年12月3日,复旦大学生物医学研究院杨力研究组在Briefings in Bioinformatics杂志上在线发表了题为“RNAlight: a machine learning model to identify nucleotide features determining RNA subcellular localization”的研究论文。该研究基于LightGBM框架开发了机器学习模型——RNAlight,RNAlight可以准确预测多类型RNA的亚细胞定位,并通过整合Tree SHAP及序列组装算法能够有效地鉴定影响RNA亚细胞定位的关键核苷酸序列特征。

该RNA亚细胞定位预测工具具有如下优势:第一、研究团队构建了一系列机器学习模型和深度学习模型来预测lncRNA和mRNA的亚细胞定位(图1)。其中,基于LightGBM(Light Gradient Boosting Machine)(3)框架的机器学习模型——RNAlight,表现出最好的预测效果,并优于其它已报道的RNA亚细胞定位预测模型。第二、通过整合Tree SHAP(SHapley Additive exPlanations)(4)及序列组装(k-mer assembly)算法,RNAlight有效地鉴定出了与RNA亚细胞定位密切相关的RNA序列特征和RNA结合蛋白。第三、该研究还应用RNAlight预测了其它类型的RNA亚细胞定位,包括核小RNA(small nuclear RNA,snRNA)、核仁小RNA(small nucleolar RNA,snoRNA)和环形RNA(circular RNA),RNAlight准确地预测出了上述类型RNA的亚细胞定位模式,提示RNAlight能够学习并鉴定出影响RNA亚细胞定位的普适性序列特征。综上,RNAlight可以完成多类型的RNA亚细胞定位预测及核苷酸序列特征鉴定,为RNA亚细胞定位及功能研究提供了新的思路和方法。
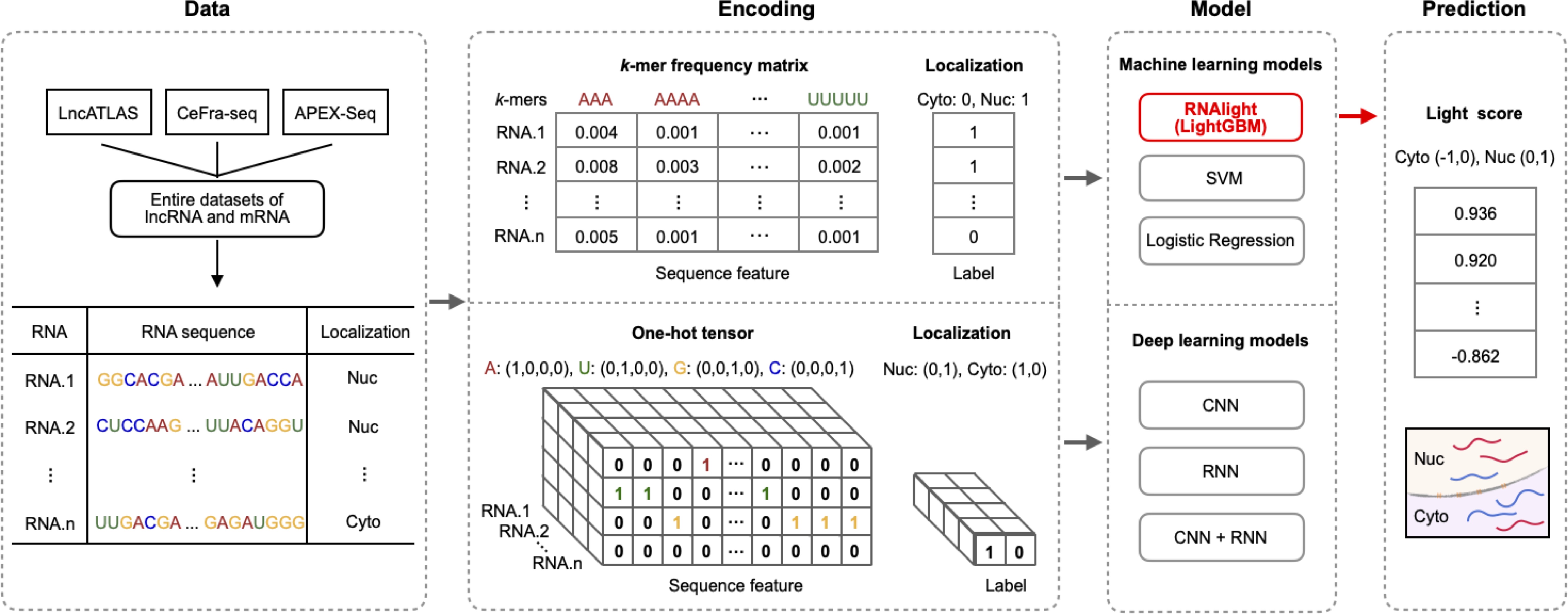
图1:RNAlight构建及预测流程示意图
复旦大学生物医学研究院杨力研究员为本文通讯作者,中国科学院上海营养与健康研究所博士研究生袁国华和博士后王滢为本文共同第一作者。中国科学院上海营养与健康研究所王光中研究员也参与了相关研究。该工作获得了国家自然科学基金委、科技部国家重点研发计划、中国博士后科学基金会、上海超级博士后激励计划等项目资助。
原文链接:
https://doi.org/10.1093/bib/bbac509
参考文献:
1.Mili S, Macara IG. RNA localization and polarity: from A(PC) to Z(BP), Trends Cell Biol 2009;19:156-164.
2.Chen LL. Towards higher-resolution and in vivo understanding of lncRNA biogenesis and function, Nat Methods 2022;19:1152-1155.
3.Ke GL, Meng Q, Finley T et al. LightGBM: a highly efficient gradient boosting decision tree, Advances in Neural Information Processing Systems 30 (Nips 2017) 2017;30.
4.Lundberg SM, Erion G, Chen H et al. From Local Explanations to Global Understanding with Explainable AI for Trees, Nat Mach Intell 2020;2:56-67.