Nature | 徐彦辉团队揭示RNA聚合酶III转录起始动态过程
RNA聚合酶(RNAPs)是转录的核心酶,负责将DNA上的遗传信息转录为RNA。在哺乳动物中,RNA聚合酶进化出三种功能分化的形式:Pol I、Pol II 和 Pol III。其中,Pol II负责转录编码蛋白质的mRNA,由于其与基因表达调控密切相关,长期以来一直是转录研究的重点对象。相比之下,Pol III主要转录多种短链非编码RNA(如5S rRNA、tRNA 和 U6 snRNA等),在蛋白质合成、RNA剪接以及细胞周期调控中发挥关键作用。
徐彦辉研究团队近年来在Pol III转录机制研究方面持续取得突破,先后解析了人源Pol III延伸复合物(elongation complex, EC)的结构与调控机制(Cell Research, 2021)、转录终止机制(徐彦辉团队Cell Discovery+Nat Comm分别揭示人源RNA聚合酶I转录调控机制及RNA聚合酶III转录终止机制),以及完整起始复合物的组装方式(Cell Research | 徐彦辉团队揭示人源RNA聚合酶III转录起始复合物的结构),为全面理解Pol III的转录过程提供了重要线索。但上述工作,以及目前几乎所有的围绕Pol III的研究工作,都没有解析PolIII转录起始的动态调控机制,我们对Pol III 转录的理解仍然停留在静止的阶段。
虽然不同类型的RNA聚合酶遵循类似的转录起始过程——包括启动子识别、转录起始复合物(pre-initiation complex, PIC)组装、DNA双链解链、转录泡形成以及RNA合成与转录延伸的转换等步骤——但不同RNAPs在调控因子依赖性、复合物组装方式及动态构象变化上存在显著差异,以适应各自所负责基因类型的转录需求。系统解析转录起始过程中的动态变化,特别是从转录起始向延伸的关键转折机制,是深入理解转录调控的核心内容,也是目前最具挑战性的研究难点之一。
2006年美国罗格斯大学Richard H. Ebright研究组首次利用单分子方法解析了细菌转录起始的动态过程,揭示了细菌RNA聚合酶在转录起始向延伸转变过程中遵循“scrunching”机制,即DNA的压缩产生应力,促使不稳定的转录起始复合物向转录延伸复合物转换,实现启动子逃逸(Science, 2006a,b)。2023年底,复旦大学徐彦辉研究员团队首次以单碱基分辨率动态揭示了哺乳动物Pol II从起始到早期延伸的全过程,指出模板链在Pol II核心通道的堆积以及NTP水解提供的驱动力是该阶段转变的关键因素(Science专家点评丨徐彦辉团队揭示转录起始连续动态全过程)。同年,比利时鲁汶大学Kalyan Das研究组与美国罗格斯大学 Smita S. Patel研究组合作报道酵母线粒体RNA聚合酶(mtRNAP)在转录起始阶段的动态变化,发现非模板链的扩张和模板链的堆积会导致转录泡崩塌和启动子逃逸,从而驱动转录起始向延伸的转换。上述工作揭示了不同物种,不同功能RNAP转录起始向延伸的转折机制,推动了对转录起始调控机制的深入理解。然而,目前对Pol III的起始调控机制了解仍较为有限,研究进展相对滞后。对于Pol III的转录起始到延伸阶段的动态转换机制,尤其是是否存在与Pol II类似的构象变化,仍知之甚少。回答这些关键问题不仅将系统补全真核转录调控网络的理解,还将为RNA聚合酶的功能差异和进化研究提供新的视角。
2025年6月4日,徐彦辉团队在Nature杂志在线发表题为Structural insights into human Pol III transcription initiation in action的研究论文。该研究重建了人源Pol III转录起始的完整动态过程,揭示了转录因子与聚合酶催化活性协同驱动Pol III由转录起始向延伸过渡的分子机制,为理解真核短链非编码RNA合成的调控提供了关键结构基础。
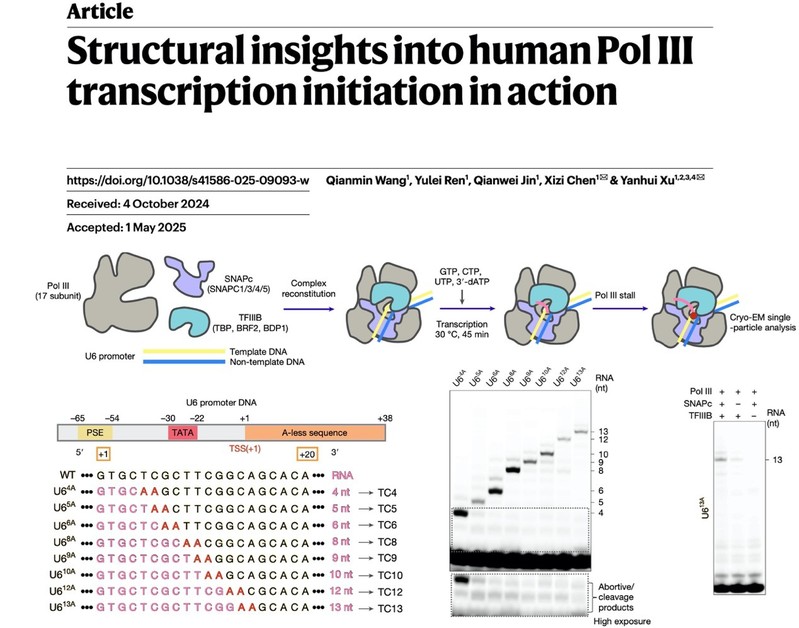
图1. Pol III 起始复合物组装与体外转录测试。
研究团队参考前期Pol II研究的方法并进行了优化,基于天然U6-1启动子序列构建了多个包含A-less cassette的启动子DNA模板。该策略在缺乏ATP的体外转录反应体系中,可将转录过程“卡顿”在起始位点下游特定位点,从而捕捉转录起始向延伸转换过程中的关键中间态。随后,研究人员将这些DNA模板与通用转录因子(GTFs),包括TFIIIB复合物、SNAPc复合物,以及Pol III共同组装,启动体外转录反应,并结合UTP、CTP、GTP和3’-dATP,获得多个不同阶段的转录复合物(transcription complex, TC),并进行冷冻电镜单颗粒分析(图1)。
转录起始到延伸的转换点:RNA长度达到6个核苷酸
分析获得的七个高分辨率Pol III-TC结构(分别停顿于转录起始位点下游4至13位核苷酸)揭示:TC4和TC5属于起始状态(initially transcribing complex, ITC),而从TC6起Pol III进入延伸状态,说明Pol III由ITC向EC的转变发生于合成第6个核苷酸时(图2)。值得注意的是,在TC5结构中观察到RNA-DNA杂合体呈现“倾斜”配对,这是由于模板链DNA处于pre-translocate状态,而新生RNA处于post-translocate状态,导致杂合体产生不对称形变。这一非经典构象由转录因子BRF2的finger结构域稳定。随着NTP持续水解,第6个核苷酸被加入至RNA链3’端,推动杂合体前移并驱动BRF2-finger结构域退出活性中心(视频1)。这一关键步骤解除对模板链DNA的限制,进而引发转录因子与Pol III之间的重构,包括BRF2核心结构域以及与其相互作用的TBP、BDP1和SNAPc的解离,实现Pol III的启动子逃逸。这一转换过程也在印迹“footprinting”实验中得到了验证。
视频1. Pol III转录起始阶段连续构象转变过程。左:显示完整转录复合物整体构象;中:为更清楚观察转录泡的形成过程和DNA与蛋白质之间的相互作用界面,隐藏了部分遮挡启动子DNA的蛋白结构;右:启动子DNA构象。其中模板链和非模板链分别显示黄色和蓝色,转录起始位点显示红色。与Pol II和线粒体RNAP相比,Pol III完成转录起始向延伸的转换所需RNA链长度更短,仅为6个核苷酸(而mtRNAP为8 nt,Pol II为10 nt)。该差异的生理意义仍需进一步探讨,推测可能与Pol III所负责的模板类型及其在高效合成短链非编码RNA中的功能要求密切相关。
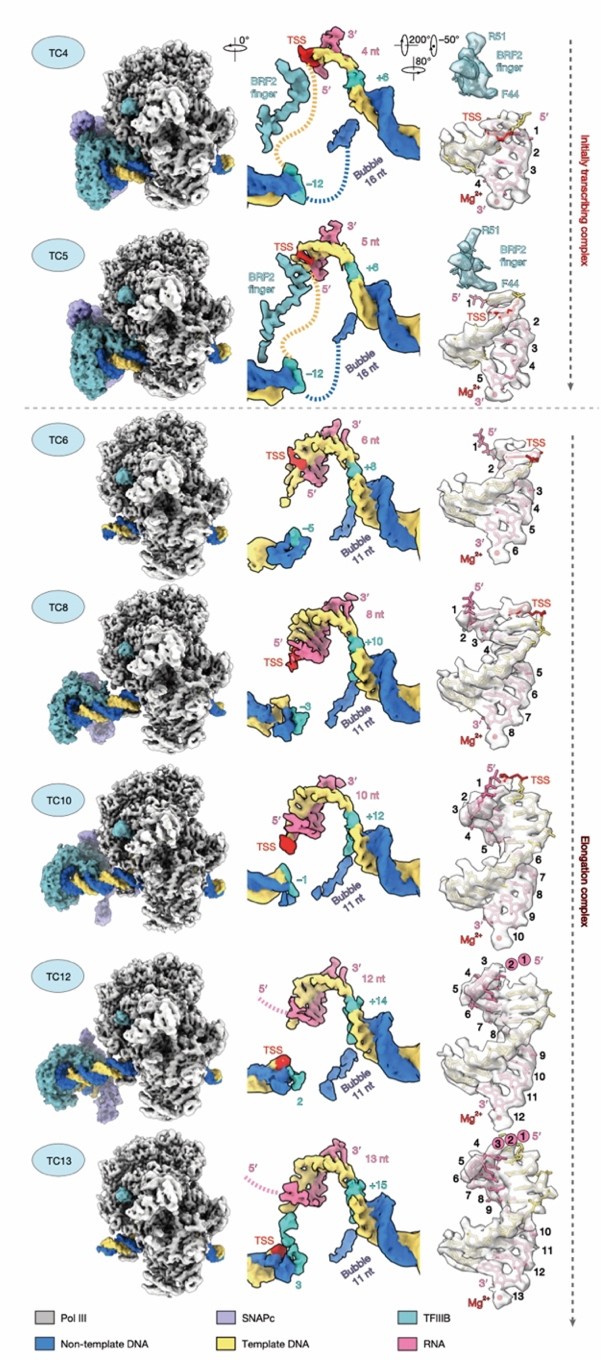
图2. Pol III转录起始动态过程:七个Pol III-TC结构示意图。
起始因子在启动子的滞留和转录再起始
另一个重要发现是关于转录“再起始”的结构证据。尽管Pol III进入延伸状态后,与通用转录因子的直接相互作用被打破,但这些因子仍稳定结合在启动子上游区域(图3)。长期以来,科学界推测Pol III起始因子在一次转录完成后不会立即脱离DNA,而是保留在启动子附近,便于迅速启动新一轮转录。本研究首次在结构层面观察到起始因子的启动子滞留状态,支持了转录再起始机制的存在(视频1)。
早期完成转录延伸转换和快速再起始机制,可能正是Pol III在执行高频率转录任务中所采用的策略。
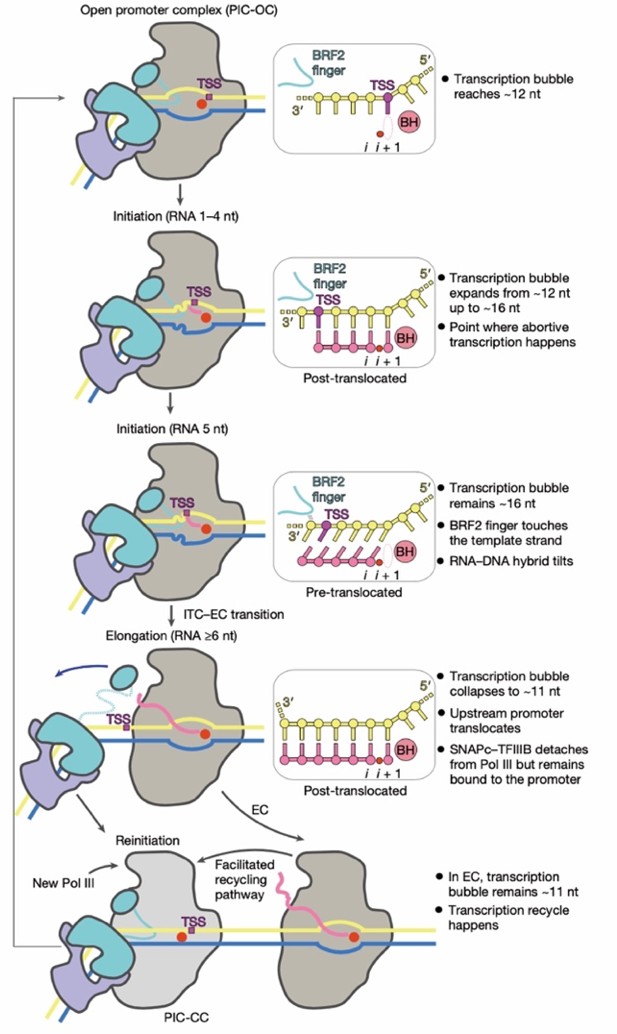
图3. Pol III转录起始模式图。
不依赖同位素的测活方法
在传统的分子生物学研究中,转录起始活性的检测主要依赖于使用同位素标记的核苷酸(如磷-32标记的UTP),这一方法已沿用超过半个世纪。然而,该技术存在诸多限制,包括同位素获取流程复杂、半衰期短、操作安全要求高等问题,严重制约了转录起始活性的常规定量分析。为克服上述瓶颈,研究团队创新性地建立了一种全新的、无需同位素的转录起始活性检测方法。该方法通过将荧光标记的小分子(pCp-AF647)高效连接至新生RNA的3’羟基,实现了对转录起始反应活性的灵敏检测。该方法灵敏度高,安全低毒,操作简便,适用于常规分子生物学实验室,大幅提升了转录研究的效率和可重复性。
复旦大学生物医学研究院青年研究员王茜敏为该文第一作者,复旦大学附属肿瘤医院/生物医学研究院/上海梧桐岛生命科学研究院研究员徐彦辉和复旦大学生命科学学院青年研究员陈曦子为该文通讯作者。
原文链接:
https://doi.org/10.1038/s41586-025-09093-w