Cell丨樊嘉/高强/张晓明/杨力合作揭示中性粒细胞的抗肿瘤潜能:抗原提呈
中性粒细胞是天然免疫的第一道防线(1),广泛参与肿瘤免疫循环、免疫编辑、免疫逃逸等重要过程(2)。同时,中性粒细胞是人体最丰富的白细胞类型,占人体白细胞的40%至70%,是肿瘤微环境的重要组成部分(2)。但是,中性粒细胞半衰期短(6-8小时)、细胞脆弱(3),这些困难导致目前鲜有中性粒细胞的大规模系统研究,他们发挥抗肿瘤还是促肿瘤的功能(4-8)是领域内争议的焦点。近年来在泛癌尺度的肿瘤微环境研究如火如荼(9-11),这一策略为系统研究中性粒细胞的功能和表型带来了新的契机。
2024年3月5日,复旦大学附属中山医院樊嘉院士和高强教授、中国科学院上海免疫与感染研究所张晓明研究员、复旦大学生物医学研究院杨力研究员合作在Cell上发表了题为Neutrophil profiling illuminates anti-tumor antigen-presenting potency的研究论文,利用自测数据(79.29%)和公共数据(20.71%),基于肝癌、胆管癌、胆囊癌等17个癌种的143例患者共计225例样本的中性粒细胞单细胞转录组,系统性揭示了人源肿瘤相关中性粒细胞的表型多样性和可塑性(如抗原提呈型、促血管新生型、炎症型等),发现HLA-DR+CD74+中性粒细胞在多个癌种和患者预后正相关,可诱导T细胞抗原特异反应并促进形成“热肿瘤”微环境,可作为潜在的免疫治疗增敏策略。

在这项研究中,研究者通过整合来自225例患者样本的中性粒细胞单细胞转录组数据,利用聚类分析发现中性粒细胞可分为10大群,如HLA-DR+CD74+抗原提呈型中性粒细胞、VEGFA+SPP1+促血管新生型中性粒细胞、CXCL8+IL1B+炎症型,这些中性粒细胞分别对应着不同的免疫表型、趋化因子特征和成熟状态。在多个癌种中,HLA-DR+CD74+中性粒细胞和更好的预后相关,而VEGFA+SPP1+中性粒细胞和更差预后相关。研究者通过综合利用4种单细胞轨迹算法、多重免疫荧光、流式细胞术等技术,表征了中性粒细胞的不同成熟状态,发现HLA-DR+CD74+中性粒细胞可能处于终末分化的成熟状态,提示了该亚群的重要作用。
因此,研究者进一步聚焦HLA-DR+CD74+中性粒细胞功能和调控机制,通过单细胞代谢通路分析、小规模代谢物体外筛选等,发现亮氨酸代谢和中性粒细胞抗原提呈功能的潜在联系:亮氨酸可上调中性粒细胞的MHC-II类抗原提呈分子HLA-DR及相关共刺激分子表达。作者进一步发现,亮氨酸可通过线粒体结构和功能重塑,并增强乙酰辅酶A生成,上调MHC-II类分子启动子区的H3K27乙酰化修饰,从而上调HLA-DR等抗原提呈分子。为了进一步研究这一群细胞的抗原提呈功能,作者发现HLA-DR+CD74+中性粒细胞可提呈肿瘤新抗原至T细胞,重塑T细胞免疫组库并增强抗原特异反应。这些结果提示了HLA-DR+CD74+中性粒细胞的代谢可塑性及治疗价值。
在此基础上,作者探索了抗原提呈中性粒细胞的抗肿瘤治疗价值。研究者发现多种类型的小鼠肿瘤中也存在CD74+抗原提呈相关的中性粒细胞亚群。在小鼠模型中,富含亮氨酸的饮食或CD74+中性粒细胞过继治疗可增强抗PD-1免疫疗法的疗效。而当抗原提呈中性粒细胞过继处理患者来源性肿瘤组织块(patient-derived tumor fragments,PDTFs)时,T细胞抗原特异反应分子和杀伤分子均显著升高。此外,基于肿瘤患者的免疫治疗测序数据分析发现,抗原提呈中性粒细胞和免疫治疗响应程度显著正相关。
综上,这项工作生成了多癌种的中性粒细胞转录组数据,深入分析了中性粒细胞癌种特异及共享的转录模式、细胞状态、代谢特征、临床相关性,聚焦HLA-DR+CD74+中性粒细胞的抗原提呈功能,探索了利用中性粒细胞抗原提呈来增敏免疫治疗的可能。
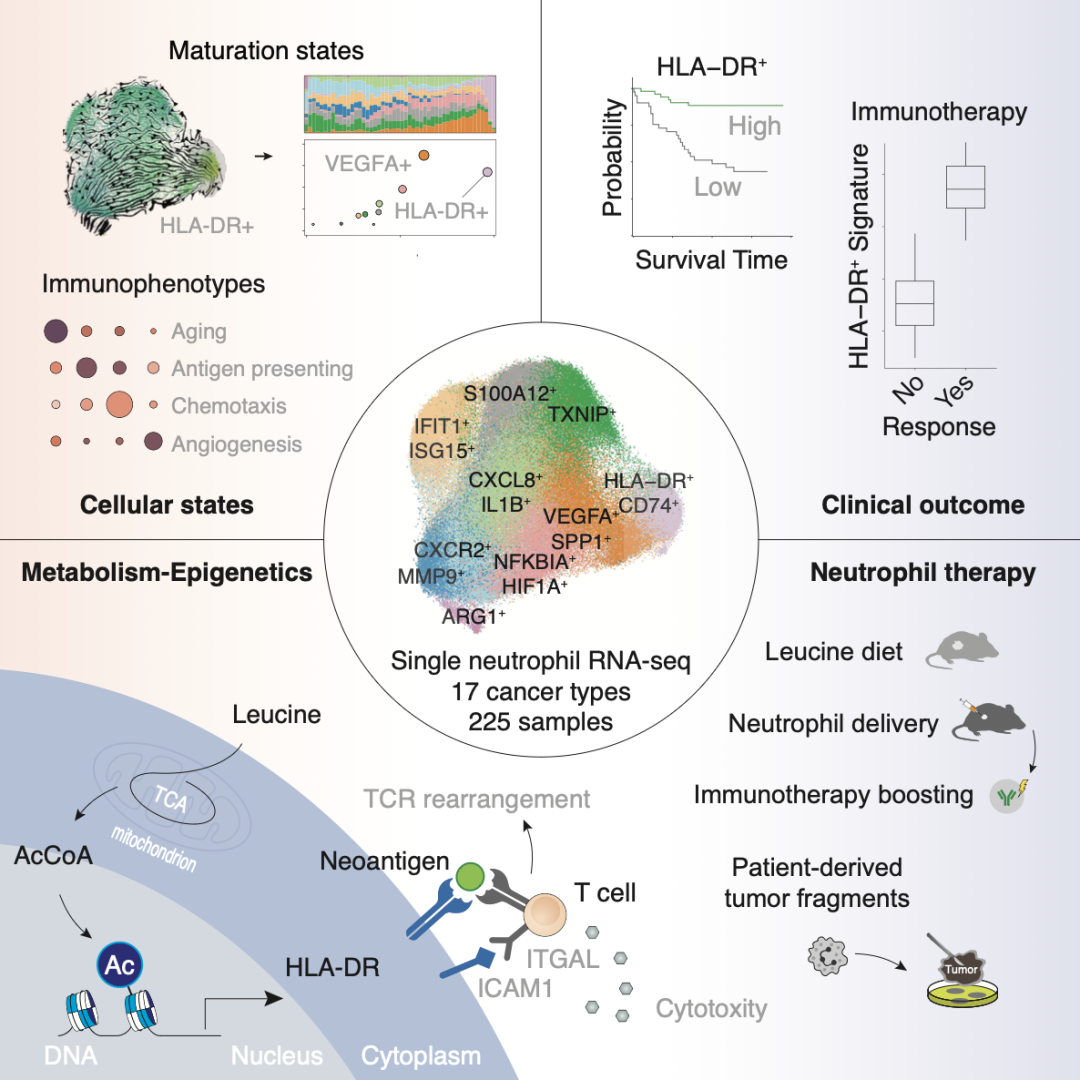
图1. 研究设计示意图。
复旦大学附属中山医院樊嘉院士和高强教授、中国科学院上海免疫与感染研究所张晓明研究员、复旦大学生物医学研究院杨力研究员为本文通讯作者。复旦大学博士生吴英成、马家强、杨旭鹏、博士后南芳、硕士生张天成为共同第一作者。本研究得到复旦大学雷群英教授、朱棣教授、美国哈佛大学麻省总院朱秀轩教授、法国巴斯德研究所Claude Leclerc教授、上海交大免疫所王峰教授和王静教授的支持。
原文链接:https://doi.org/10.1016/j.cell.2024.02.005
参考文献:
1. Burn, G.L., Foti, A., Marsman, G., Patel, D.F., and Zychlinsky, A. (2021). The Neutrophil. Immunity 54, 1377-1391. 10.1016/j.immuni.2021.06.006.
2. Hedrick, C.C., and Malanchi, I. (2022). Neutrophils in cancer: heterogeneous and multifaceted. Nat Rev Immunol 22, 173-187. 10.1038/s41577-021-00571-6.
3. Summers, C., Rankin, S.M., Condliffe, A.M., Singh, N., Peters, A.M., and Chilvers, E.R. (2010). Neutrophil kinetics in health and disease. Trends Immunol 31, 318-324. 10.1016/j.it.2010.05.006.
4. Ng, M.S.F., Kwok, I., Tan, L., Shi, C., Cerezo-Wallis, D., Tan, Y., Leong, K., Calvo, G.F., Yang, K., Zhang, Y., et al. (2024). Deterministic reprogramming of neutrophils within tumors. Science 383, eadf6493. 10.1126/science.adf6493.
5. Xue, R., Zhang, Q., Cao, Q., Kong, R., Xiang, X., Liu, H., Feng, M., Wang, F., Cheng, J., Li, Z., et al. (2022). Liver tumour immune microenvironment subtypes and neutrophil heterogeneity. Nature 612, 141-147. 10.1038/s41586-022-05400-x.
6. Cui, C., Chakraborty, K., Tang, X.A., Zhou, G., Schoenfelt, K.Q., Becker, K.M., Hoffman, A., Chang, Y.F., Blank, A., Reardon, C.A., et al. (2021). Neutrophil elastase selectively kills cancer cells and attenuates tumorigenesis. Cell 184, 3163-3177 e3121. 10.1016/j.cell.2021.04.016.
7. Hirschhorn, D., Budhu, S., Kraehenbuehl, L., Gigoux, M., Schroder, D., Chow, A., Ricca, J.M., Gasmi, B., De Henau, O., Mangarin, L.M.B., et al. (2023). T cell immunotherapies engage neutrophils to eliminate tumor antigen escape variants. Cell 186, 1432-1447 e1417. 10.1016/j.cell.2023.03.007.
8. Kalafati, L., Kourtzelis, I., Schulte-Schrepping, J., Li, X., Hatzioannou, A., Grinenko, T., Hagag, E., Sinha, A., Has, C., Dietz, S., et al. (2020). Innate Immune Training of Granulopoiesis Promotes Anti-tumor Activity. Cell 183, 771-785 e712. 10.1016/j.cell.2020.09.058.
9. Cheng, S., Li, Z., Gao, R., Xing, B., Gao, Y., Yang, Y., Qin, S., Zhang, L., Ouyang, H., Du, P., et al. (2021). A pan-cancer single-cell transcriptional atlas of tumor infiltrating myeloid cells. Cell 184, 792-809 e723. 10.1016/j.cell.2021.01.010.
10. Zheng, L., Qin, S., Si, W., Wang, A., Xing, B., Gao, R., Ren, X., Wang, L., Wu, X., Zhang, J., et al. (2021). Pan-cancer single-cell landscape of tumor-infiltrating T cells. Science 374, abe6474. 10.1126/science.abe6474.
11. Kinker, G.S., Greenwald, A.C., Tal, R., Orlova, Z., Cuoco, M.S., McFarland, J.M., Warren, A., Rodman, C., Roth, J.A., Bender, S.A., et al. (2020). Pan-cancer single-cell RNA-seq identifies recurring programs of cellular heterogeneity. Nat Genet 52, 1208-1218. 10.1038/s41588-020-00726-6.