Brief Bioinform丨杨力团队与合作者开发蛋白质亚细胞定位预测的新型“文生图”多模态模型-deepGPS
蛋白质的功能行使与其亚细胞定位密切相关,因此鉴定蛋白质的亚细胞定位对于理解其在不同生理和病理条件下的功能至关重要。除了利用实验直接观测蛋白质的亚细胞定位,近年来针对蛋白质亚细胞定位预测的计算生物学工具也相继被开发出来。按照其输入和输出类型的不同,上述计算生物学工具主要可分为两类:一类是以DeepLoc为代表的基于蛋白质序列输入、生成定位文本标签(text-to-text)的预测模型;另一类是以Cytoself为代表的自监督蛋白质定位聚类模型,该模型利用变分自编码器对原始蛋白定位图像进行重构(image-to-image),以学习蛋白质定位模式。然而,前者仅输出文本标签,难以直观展现蛋白质的定位分布,而后者虽能解析已知蛋白的定位模式,但无法预测未知蛋白的定位图像。
2025年4月12日,复旦大学生物医学研究院杨力研究组与上海人工智能实验室董楠卿研究组合作在Briefings in Bioinformatics杂志在线发表了题为Deep Generative Model for Protein Subcellular Localization 的研究论文。该研究基于ESM2蛋白质大语言模型及U-Net框架开发了具备多模态处理能力的生成式深度学习模型deepGPS,deepGPS能够接收蛋白质序列及细胞核图像作为输入,并生成蛋白质定位的文本标签及分布图像,是一种支持蛋白质亚细胞定位预测的新型“文生图”(text-to-image)多模态模型。研究团队还构建了开放的openGPS平台(https://bits.fudan.edu.cn/opengps/),支持蛋白质亚细胞定位的在线预测及实验图像整合,助力蛋白质亚细胞定位与功能研究。

在deepGPS模型中,研究团队设计了一个整合文本(text)和图像(image)的预测体系,其包括两条并行的计算路径:第一条路径基于蛋白质序列,通过ESM2和MLP层(Multilayer Perceptron)提取序列特征,并将其转换为序列隐向量(sequence latent vector),随后输出蛋白质定位预测的文本标签(图1,下方);第二条路径以细胞核图像为参考,利用U-Net进行特征提取,细胞核图像首先经过下采样,转换为图像隐向量(image latent vector),然后通过注意力机制融合序列隐向量与图像隐向量,最终经过上采样生成符合给定细胞核分布的蛋白质定位图像(图1,上方),进而实现从蛋白质序列到定位图像的从头预测(文生图)。利用deepGPS框架,研究团队基于OpenCell和Human Protein Atlas数据库中的蛋白质定位注释及图像数据,分别训练了适用于HEK293T和U2OS细胞的deepGPS细胞特异性模型。在亚细胞定位预测的二分类(Cytoplasm和Nucleoplasm)任务中,deepGPS在准确性、敏感性、F1 score及AUPRC等指标上均优于现有的text-to-text模式的蛋白定位预测工具。重要的是,尽管存在数据量不足的因素,deepGPS在四分类(Cytoplasm、Nucleoplasm、ER和Vesicles)及OpenCell数据所涉及的所有亚细胞定位任务中表现出了较好的预测/图像生成效果。最后,团队开发了openGPS平台,期望通过在线预测和实验数据搜集,促进蛋白质亚细胞定位及功能研究。
综上,deepGPS的构建不仅为蛋白质定位预测提供了创新方法,也是计算生物学领域中“文生图”多模态研究的一次重要探索。这项工作是杨力研究组前期开发针对多类型RNA亚细胞定位预测机器学习text-to-text模型(Yuan et al., Brief Bioinform, 2023)的拓展,在方法上实现了创新。
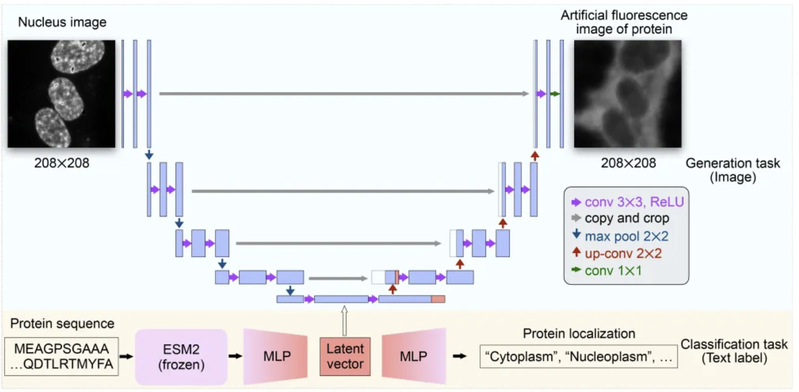
图1:deepGPS构建及预测流程示意图
复旦大学生物医学研究院杨力研究员和上海人工智能实验室科学智能中心董楠卿副研究员为本文共同通讯作者。中国科学院上海营养与健康研究所博士研究生袁国华、上海人工智能实验室和复旦大学信息科学与工程学院联合培养博士研究生李劲哲、中国科学院计算技术研究所博士研究生杨泽君和复旦大学生物医学研究院博士研究生陈尧琦为本文共同第一作者。该工作得到上海人工智能实验室欧阳万里教授和复旦大学信息科学与工程学院陈涛教授的大力支持和技术指导。
原文链接:https://doi.org/10.1093/bib/bbaf152